Published at 2021-06-16 11:33
Author:zhixy
View:4299
所有生命共享一个祖先。支持该理论的一个重要例证是:存在于所有生命体中的"Universal"基因。我们可以在种内或种间找到非常相似的基因,进而推测它们是进化上相关、共享同一祖先的,即同源的 (homologous)。
确定同源基因是非常有趣而且重要的,因为这是回答进化相关问题(如:哪些是保守的?哪些是发生变化的?)的第一步。此外,由于从实验上描绘基因的特征仍然是费时费力的,所以评估进化关系成了一种在不同物种间获得基因属性内延和外延的重要方式。
确定同源关系的一个重要环节是,进一步区分同源基因是以何种方式关联在一起的,这直接导致了同源关系的不同亚型。由物种分化 (speciation) 而导致的同源,称为直系同源 (orthologs);产生于基因复制 (gene duplication) 的同源关系,称为旁系同源 (paralogs);而如果与基因组复制 (genome duplication) 或基因组同源多倍体化 (homopolyploidization)* 有关,则称为ohnologs;通过杂交,而后基因组异源多倍体化 (allopolyploidization*) 产生的,称为homoelogs;由横向基因转移 (lateral gene transfer)* 导致的,称为xenologs***。
在以推断物种系统发育为主要目的的系统发育基因组学 (phylogenomics) 中,直系同源有着无可替代的重要性。
ohnologs, 为纪念日本遗传学家Susumu Ohno (大野乾)。
直系同源 (orthologs) 最早由Walter M. Fitch于1970年定义。
"It is not suffcient, for example, when reconstructing a phylogeny from amino acid sequences that the proteins be homologous. [. . . ] there should be two subclasses of homology. Where the homology is the result of gene duplication so that both copies have descended side by side during the history of an organism (for example, α- and β- hemoglobin) the genes should be called paralogous (para = in parallel). Where the homology is the result of speciation so that the history of the gene reflects the history of the species (for example α hemoglobin in man and mouse) the genes should be called orthologous (ortho = exact). Phylogenies require orthologous, not paralogous, genes."
虽然上述定义是比较有远见而且清晰的,然而仍然有一些隐含意义和复杂性经常在文献资料中造成误解和矛盾。
首先,需要强调是Fitch定义的直系同源和旁系同源描述的是两个基因之间的关系,与导致这两个基因产生的最初进化事件的类型直接相关。这就意味着,发生在物种分化之后的基因复制,将不再影响两者关系的确定。这种复制将导致一个基因在另一物种中存在多个直系同源对象。换句话说,直系同源描述的不仅是一对一关系,也可描述一对多、多对一、或者多对多的关系。
再者,该定义并未考虑相关基因在基因组上的位置。举例说明,假设有一个起源于哺乳动物共同祖先的基因,传至人类仅留下了一个子代基因,而在啮齿动物中发生了复制,即一个正本和一个复本。那么,在老鼠基因组中,这个基因的正本会保留在原始位置上,而它的复本会移动到基因组的其它位置上。啮齿动物中互为旁系同源关系的基因正本和复本,与人类中的同源基因又都为直系同源关系。为区别此类直系同源关系,Deway 2011年建议了位置直系同源 (positional ortholog),特指位于保守基因座位上的直系同源基因。
在Fitch列举的例子中,旁系同源关系 (α- 和 β- hemoglobin) 存在于同一个物种中;直系同源关系 (人类和老鼠的α-hemoglobin) 存在于不同物种中。那么来自不同物种的两个基因是否也可呈现旁系同源关系呢?答案是肯定的。比如老鼠的α-hemoglobin和人类的β-hemoglobin。但是反过来,同一物种的两个基因是否会呈现直系同源关系呢?这就要考虑以下场景的可能性,即由物种分化导致的两个基因最终出现在同一个物种的可能性。虽然不常见,但还是有可能发生的 (通过横向基因转移或杂交)。但通常这类情形,应该分别用不同的术语来定义:xenologs和homoeologs。
Fitch的原始定义描述的是基因的成对关系 (pairwise relationships),实际场景中往往需要在多个物种中应用这些概念。但是这些概念一般化并不是那么直接了当,因为直系和旁系同源关系不具备传递性。假设A基因是B基因的直系同源基因,而B基因又是C基因的直系同源基因,仅基于此并不能得出A基因和C基因也互为直系同源关系,例如下图所示的人insulin基因INS和老鼠的insulin基因Ins1和Ins2。
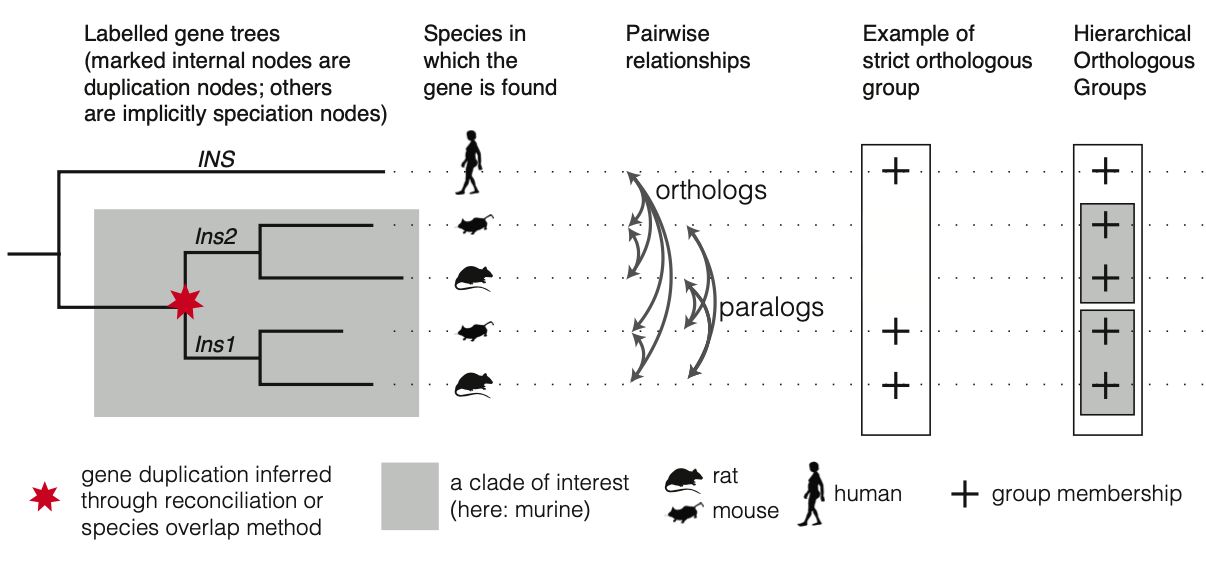
成对关系会依基因和物种的数量呈二次增长。此外,正如所看到的,在基因组之间或跨物种之间没有直接的成对关系推断。因此,为了在多物种的场景下表达和解释基因间的成对关系,促生了直系同源群 (orthologous groups) 的概念。当前,有两类不同的直系同源群。一种称为严格直系同源群 (strict orthologous groups, sOGs);另一种称为分层直系同源群 (hierarchical orthologous groups, hOGs)。
sOGs要求所有基因两两之间都为直系同源关系。实际上,这里要求的是一对一的直系同源关系,因为只有一对一的关系是可以传递的。更一般地,只要产生的基因同源群不包括任何旁系同源关系,这种同源群就有可能跨越基因复制事件。例如上图所示的,人INS基因、小鼠Ins1基因和大鼠Ins1基因就构成了一个严格直系同源群。
hOGs目的是在一个特定的祖先物种中,描述一群从一个共同祖先基因遗传下来的基因。在insulin的例子中,由于人类INS、小鼠Ins1和小鼠Ins2都来自所有哺乳动物共同祖先的一个基因,因此它们在该水平上 (即哺乳动物) 属于一个共同的分层同源群。相比之下,Ins1和Ins2在小鼠共同祖先之前由复制产生,这两个基因在鼠科动物水平上属于不同的分层同源群。因此,hOGs是依据特定的系统发育分支来定义的。而且,我们还可以看到它们的层次性质,即定义在更深层次分支上的hOG包含了定义在其后代分支上的多个hOGs。hOGs的定义类似于过去的基因“亚家族”概念,用来描述具有共同祖先的基因家族成员的亚集。
作为同源群的替代方案,也可以使用有根基因树确定同源关系,有根基因树的内部节点被标记为物种形成或基因复制节点(甚至可能是更多类型的进化事件)。这类树通常被称为“标记”或“调和”基因树。在基因树上,现存基因之间的成对的直系同源和旁系同源关系,可以从与它们共同祖先相关的标记中推断出来:如果最后共同祖先是一个物种形成节点,这两个基因则是直系同源的;如果是基因复制,那么它们就是旁系同源的。
同样地,可以从感兴趣的分类范围相对应的物种形成节点的分支中得到分层同源群。因此,标记基因树可以呈现所有的直系同源和直系同源群的信息。此外,基因树传递了基因复制的顺序,并通过分支长度量化序列分歧的程度。
直系同源关系推断方法通常分为两大类:基于树的方法和基于图的方法。
顾名思义,基于树的方法,在其算法的某个阶段将明确地推断出基因树。相比之下,基于图的方法则避开了树推断,而是以成对的方式比较序列,并构建一个以基因为顶点和序列相似度量作为边权重的图 (graph)。
基于树的直系同源推断方法为一群同源序列重建一棵基因树,然后推断出该树的每个内部节点所代表的进化事件类型。为推断内部节点上的进化事件,传统的方法是执行“基因树/物种树的调和”,这可以在简约或似然法框架中实现。或者,内部节点的标记可以通过物种重叠 (species overlap) 的方法确定,内部节点之下的多个子树中具有相同的物种,该内部节点将被标记为基因复制节点。因此,物种重叠法不需要或假设任何物种树。或者,更确切地说,它注重的是一棵完全未解的物种树。因此,它依赖于每个基因复制所产生的两个拷贝保留在至少一个物种中,这在实践中也经常如此。与传统的基因/物种树调和方法相比,该算法对基因树的拓扑多样性具有更加强的鲁棒性,该方法倾向于引入基因复制事件来解释与规范物种树的任何背离。
基于图的方法是基于物种内部和物种之间的基因对之间的比较。它们都是基于:对于两个物种之间的基因对,同源基因往往是分化最小的序列对。这是因为在与这两个物种相关的物种形成事件之前,直系同源基因是相同的基因,而旁系同源基因是早期基因复制的结果,因此有更多的时间来分化。
基于这一理解诞生了第一个大规模的直系同源预测方法,即“双向最优匹配” (bidrectional best hit, BBH) 法,它考虑的是互为比对得分最高的序列对,在系统发育距离意义下的对应概念是“相互最短距离” (reciprocal shortest distance)。
然而,BBH和RSD并不能很好地处理多对多的直系同源关系,从而导致假阴性结果。为了解决这个问题,Inparanoid算法提供了一种识别多对多直系同源关系的方法。此外,有差别基因丢失可能会导致BBH和RSD失效,因为当相应的直系同源基因在两个物种中都缺失,会导致它们的旁系同源物被错误地识别为直系同源基因。OMA算法引入了第三方物种,而第三方物种可能保留了这两个副本,因此可以作为“非直系同源关系的见证”。
BBH和RSD的另一个局限是,它们并没有明显地推广应用到成群直系同源关系确定。COGs数据库率先使用两对同源物的“三角关系”来建立多物种的直系同源组 (辅以手工矫正)。OrthoMCL则使用马尔可夫聚类法来代替。然而,OrthoMCL的一个问题是,基因同源群的粒度取决于“膨胀参数 (inflation parameter)”的选择,这使得其结果难以解释。
自系统发育学起源以来,解析生命之树一直是进化生物学中所有系统层面的普遍问题之一。从细菌到真核生物,从古菌到后生动物,人们为理解生物体之间的进化关系付出了巨大的努力。
推断物种树的系统发育信息最开始来源于形态特征。这些特征首先根据分类学比较将这些特征归类为同源或非同源特征,然后归类为祖先或衍生特征;最后根据简约原则推断出系统发育关系。随着分子生物学技术的出现,努力的方向转向使用分子标记,它们被对齐、串联 (如果使用几种标记分子),并用于重建系统发育历史。这种方法只有在对齐序列彼此直系同源的情况下才能提供合理的结果,因为直系同源定义了物种形成节点,这构成了物种树中唯一的节点类型。如果包含的一些序列具有旁系同源关系,那么一些重建的节点将确定代表基因复制,由此产生的拓扑结构将会于期望的物种树存在结构偏差。
最初,分子系统发育的实验设计包括识别感兴趣的谱系中高度保守的区域,通过聚合酶链反应 (PCR) 用特定的引物扩增。由于相同的标记分子,也就是直系同源基因,是从每个感兴趣的物种中特异性的测序获得的,因此没有必要去寻找直系同源基因。然而,诸如旁系同源基因的交叉扩增、缺少直系同源基因时的非特异性扩增、或隐性的旁系同源等常见的问题,可能使物种树重建过程复杂化,其根源在于未能获得完全直系同源基因的序列数据集。得益于高通量测序技术,完整的(或几乎完整的)基因组、转录组数据的不断涌现,我们原则上可以在几乎任何标记分子中进行选择。这时就需要从基因组数据集中推断出直系同源基因,这也成为了准确重建物种树的关键。
在过去的十年里,来自非模式生物体的基因组和转录组序列激增。通常情况下,包括转录组和低覆盖度基因组的系统发育基因组数据集是不完整的,时常包含错误和未解决的亚型。这些特征可能会严重有悖于一些直系同源关系推理方法背后的假设。因此,不同的直系同源推断方法可以导致非常不同的系统发育分析结果。尽管如此,在重建物种树的典型系统发育学分析中,并不常考虑直系同源推断的作用。相反,方法上的讨论主要集中在系统发育重建参数的问题上,例如所选择的替代模型,或着诸如数据缺失、组成异质性或谱系分选不完全等混合因素的效应。围绕栉水球或海绵作为生命动物树中最早的分支门的位置的激烈争论或许就是最好的例证。
直系同源基准化分析法 (orthology benchmarking) 要求关于基因树和物种树的精选信息。因此,当目标是推断物种树时,不同直系同源推断方法的比较是评估结果拓扑鲁棒性的最合适替代方案。然而,很少有研究比较通过不同方法推断的直系同源基因集的变化,以及如何影响物种树的重建。
选择一个合适的直系同源推断软件是特别重要的,特别是在复杂的,存在基因和基因组复制的进化场景中。没有明确考虑这些复制事件的推断方法,如OrthoMCL,被认为在植物相关的分析中存在潜在问题,因为它们倾向于将基因家族分隔而不是保留其结构。因此,在这一具有挑战性的系统发育场景中,建议采用其他能够更好地解释基因复制的方法,例如OrthoFinder,OMA,PhylomeDB。
无论选择哪个软件,包含旁系同源序列都可能会导致不同的结果。在某些情况下,如在浅层的系统发育中 (例如在目、属等的水平上),物种树的重建可能不受旁系同源基因的影响,只要它们足够最近,足以形成单系分支。甚至在一些情况下,旁系同源序列被证明作为系统发育的附加位点信息是有用的。
直系同源是系统发育组学的一个基本概念。它的定义、含义和令人生畏的推断方法在基因组学的早期导致了一些混乱。然而,挑战依然存在。在两个以上物种的背景下,同源群的概念在文献中仍然不精确;我们还没有对成对直系同源和成群直系同源达到相同水平的理解。方法之间的比较也主要集中在成对直系同源推断上。但是,基因组系统发育树推断依赖成群直系同源,最近的几项研究观察到从不同直系同源推断方法获得的树存在实质性差异。因此,为了解决困难的系统发育,有必要更好地理解和描述直系同源对树推断的影响。
全文翻译自: Rosa Fernández, Toni Gabaldón, and Christophe Dessimoz (2020). Orthology: definitions, inference, and impact on species phylogeny inference. In Scornavacca, C., Delsuc, F., and Galtier, N., editors, Phylogenetics in the Genomic Era, chapter No.2.4, pp.2.4:1–2.4:14.
