Published at 2020-04-29 09:42
Author:zhixy
View:3882
Graphlan 是一个可视化进化树和基于分类等级绘制层级分类树的工具,基于Python2.7,可通过conda安装conda install -c bioconda graphlan,或下载源代码使用。
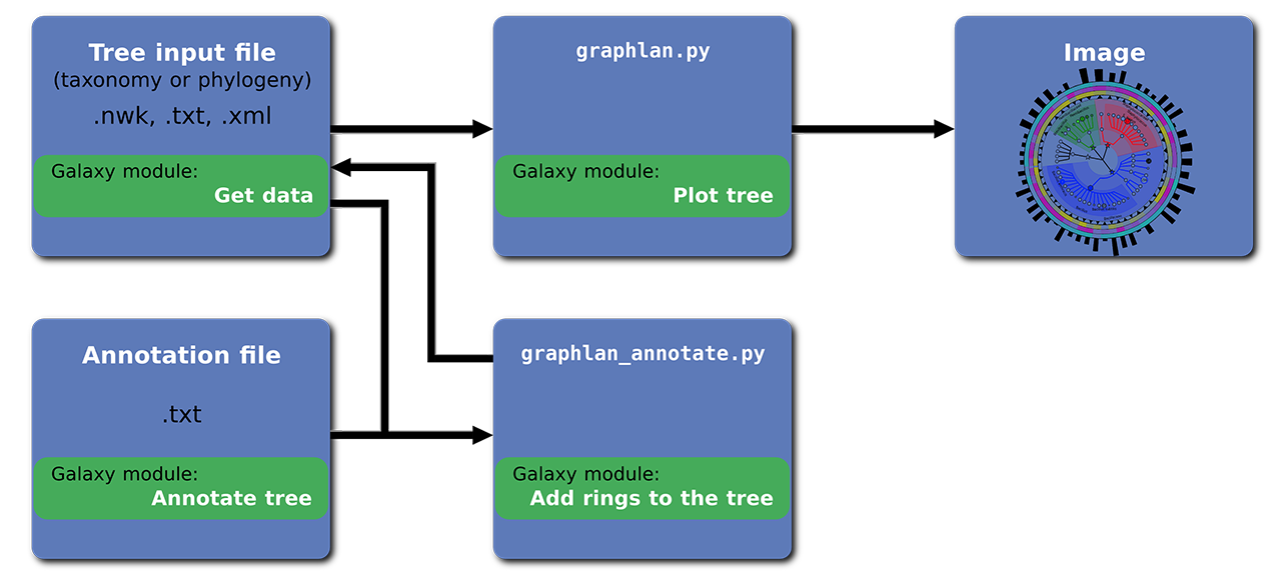
由于Graphlan基于python2.7,conda安装时,可能需要指定安装环境的python版本。
如上图所示Graphlan有两种输入文件:
和两个python脚本:
显然,Graphlan的使用关键在于graphlan_annotate.py脚本的掌握。
通常由建树软件,如IQ-TREE,RAxML等,生成的进化树格式多为Newick格式,因此可直接作为graphlan_annotate.py的输入文件。
或者利用Biopython中的Phylo模块将Newick格式树转为PhyloXML格式的树。
PhyloXML是专门针对系统发育树而设计的XML,是一种用于标记电子文件使其具有结构性的标记语言,XML的先驱之一即HTML网页标记语言。
graphlan_annotate.py实际上是在xml格式树文件的基础上,添加了各种注释标记,例如针对树节点的标记,以及树外围圈形热图等。 下图以分类层级关系(可理解为无枝长的进化树)输入文件为例,显示了Graphlan的可视化树型结构的逻辑过程。
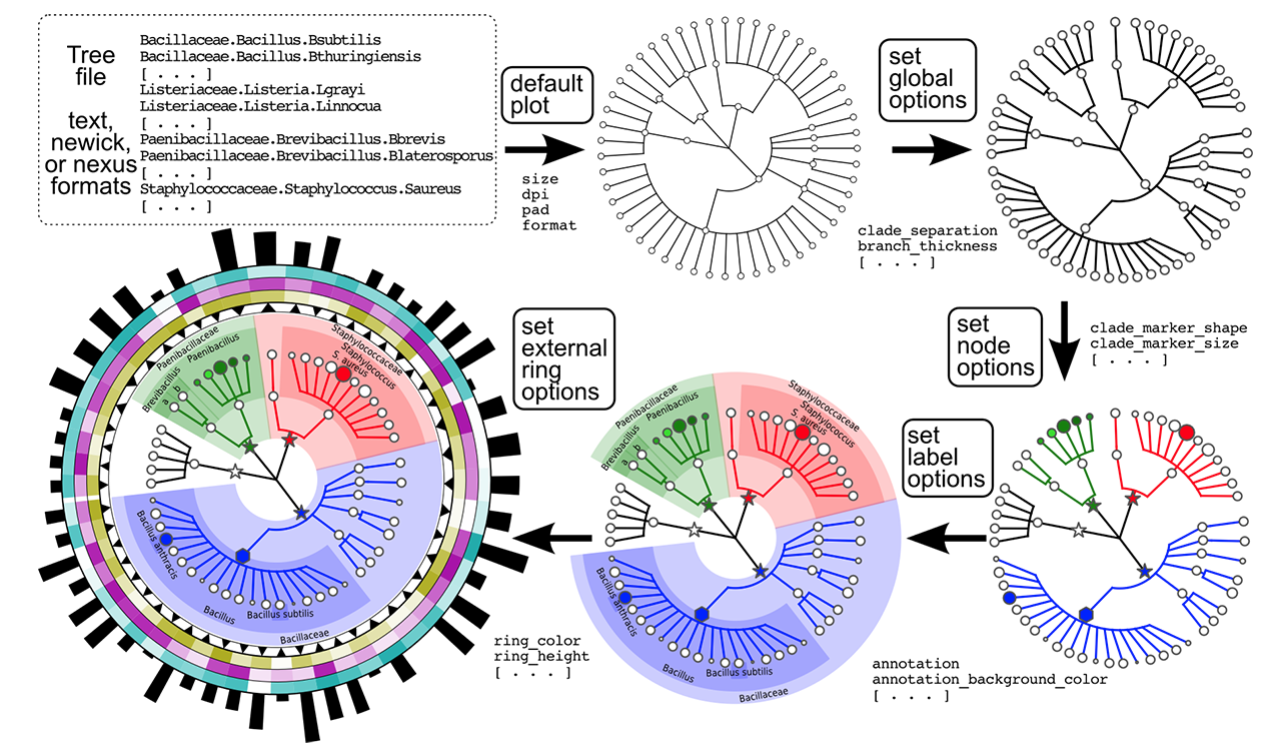
分类层级关系输入文件中的层级关系与Lineage格式数据一致,即分类单元按照层级高低自左向右按顺序排列,并用.分隔。
将层级关系文件输入graphlan.py生成的图形,即不加任何注释的默认图(default plot)。
注释标记信息写入Annotation file,通过graphlan_annotate.py可以加到默认图上。首先,全局图形选项(global options)可定义核心树形的显示效果, 如树的扇形角度、树枝粗细、子分支的分隔程度等。然后,通过指定树节点的名称,并附以节点选项(node options)的设置,可定义不同分枝的显示效果。再者, 通过指定树节点的名称,并附以标记选项(label options)的设置,可定义不同分枝的文字注释、背景色等。最后,通过指定树末端节点的名称, 附以外圈选项(external ring options)的设置,可定义多层丰富的注释信息。
因此,逻辑上Graphlan对树的可视化,可分为三个方面:
理解此点,对掌握Graphlan的使用有很大帮助。
通过graphlan_annotate.py添加的注释信息,可同分步进行,例如按上述逻辑,三步添加不同的注释信息。也可通过一个注释文件,把所有的注释信息一次添加到树上。
为树的各节点定义属性,须首先指定节点的名称,如不指定节点的名称,则对所有节点都有效。在注释文件中的写法如下:
[clade_name{|*|^}] graphical_tree_option graphical_tree_option_value
其中graphical_tree_option包括:
Graphlan支持的树节点形状包括:
树节点名称写法的解释: * 包含该节点及该节点所属的所有子节点; 包该节点及该节点所属的所有末端节点; ^ 该节点(不含其本身)下属所有末端节点。不加以上三种标记则仅对该节点有效。 另外,在注释文件中的写法,应为clade_name / clade_name / clade_name* / clade_name^
为节点及其代表的子分枝添加文字或背景颜色注释,同样需要首先指定节点名称,然后定义相关属性,包括:
从核心树图向外,Graphlan可添加多层圈形,类似热图的注释信息,如通过打标记显示定性数据,或通过定义圈的高度来显示定量信息。在注释文件中的写法如下:
[clade_name] ring_option ring_level ring_option_value
ring_option包括:
ring_level定义了圈层的位置,核心树图向外第一圈ring_level=1
示例:
clade_name ring_shape 1 v
注意:注释文件中节点名称、各属性、及取值,都使用制表符
tab键分隔的。
Graphalan的使用,主要是要根据设计需求,按照其格式规范编写好注释文件。通常情况下,需要使用Graphlan可视化进化树或分类层级信息的, 都是较大的树,或层级结构中含有较多的数据。特别是对外圈层的设置,需要写入注释文件的内容很多,手工输入是不现实的。因此需要借助其他工具 来批量生成注释内容。这需要我们对进化树的格式解析、格式的转换、以及Graphalan的注释格式规范都有较好的理解。
Graphlan原代码包中提供了不少实例,推荐按照guide文件夹中提供的数据,逐个实践相关方法。
