Published at 2020-04-18 10:26
Author:zhixy
View:2693
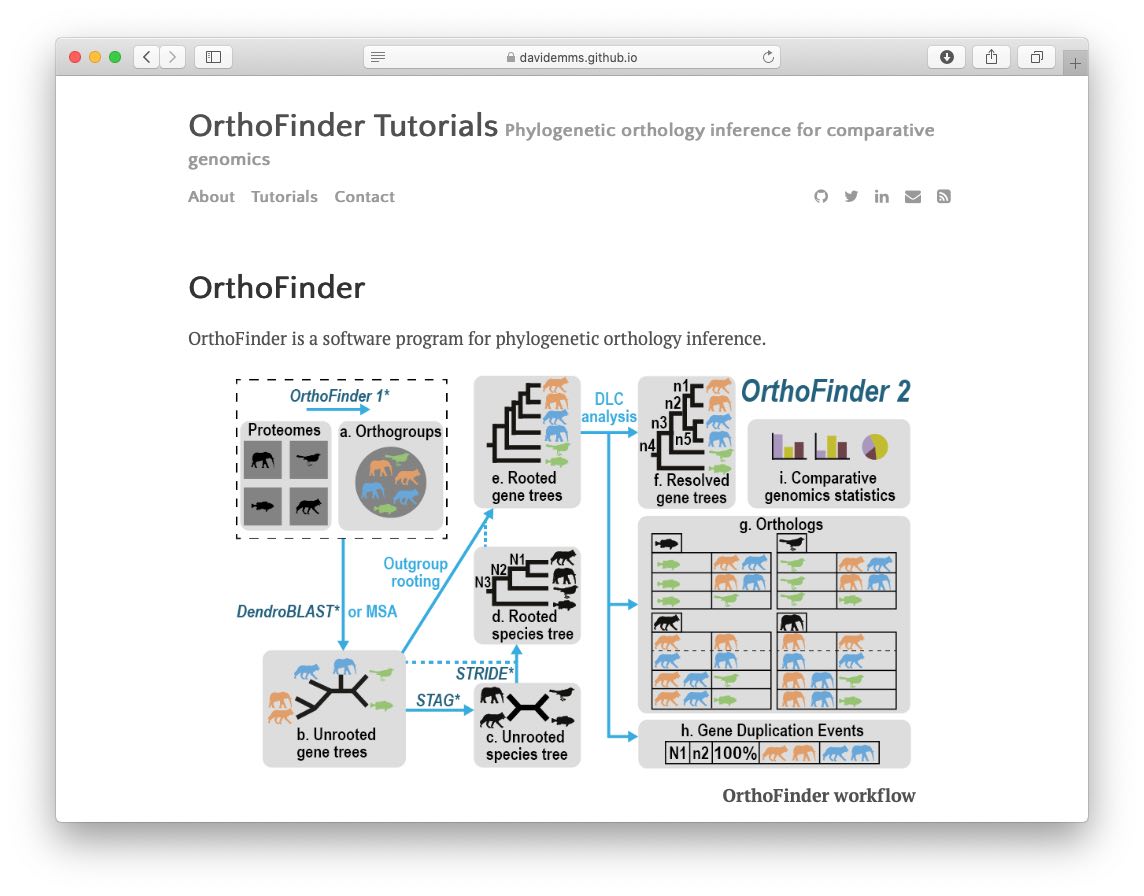
以下内容基于OrthoFinder 2.2.6
在各种安装方法中,推荐通过conda安装。首先为orthofinder创建独立的conda环境,并在该环境下安装orthofinder。
[user@server ~]# conda create -n orthofinder
[user@server ~]# conda activate orthofinder
[user@server ~]# conda install -c bioconda orthofinder
试运行orthofinder:
[user@server ~]# orthofinder
OrthoFinder version 2.2.6 Copyright (C) 2014 David Emms
SIMPLE USAGE:
Run full OrthoFinder analysis on FASTA format proteomes in <dir>
orthofinder [options] -f <dir>
Add new species in <dir1> to previous run in <dir2> and run new analysis
orthofinder [options] -f <dir1> -b <dir2>
Orthofinder的命令行提示文档已明确支出,其主要的的输入数据为FASTA format proteomes。因此,在运行orthofinder前需要准备好所需数据。
将所有受试基因组的注释结果的.faa文件(预测蛋白质序列文件),拷贝至同一文件夹下,如dir1。
在单个
.faa文件中,每条蛋白质序列的locus tag,在全部.faa文件中须保持唯一。因此建议locus tag按genome_id|locus_id的方式编码。
Orthofinder的运行大致分为以下前后衔接的环节,分别可通过五个参数停止在相应环节。
[user@server ~]# orthofinder -op -f <dir1> # 两两基因组的BLAST比对
[user@server ~]# orthofinder -og -f <dir1> # 计算orthogroups
[user@server ~]# orthofinder -os -f <dir1> # 为orthogroups提取序列
[user@server ~]# orthofinder -oa -f <dir1> # 针对各orthogroups进行MSA多序列比对
[user@server ~]# orthofinder -ot -f <dir1> # 针对各orthogroups计算基因树
在Phylogenomics分析中,通常我们仅借助Orthofinder进行基因家族的计算,即得到orthogroups结果即可停止(-og),或者在得到各orthogroups
的多序列比对结果后停止(-oa)。
对于大数据集合,基因家族分析是非常耗时的,一旦发现数据准备不足,重头再来显然是不能接受的。因此orthofinder提供了在原有计算结果上追加 数据的功能。
[user@server ~]# orthofinder -op -f <dir2> -b <dir1> # dir1是上一轮计算的输入文件夹,dir2是追加的输入数据。
同时orthofinder支持多线程计算(-t 16),可大大缩短运算时间。
-S设定序列搜索的第三方程序,可选blast, blast_gz, diamond;
-A设定多序列比对第三方程序,可选muscle, mafft;与-M msa联合使用;
-T设定构建基因树的第三方程序,可选iqtree, fasttree, raxml;与-M msa联合使用;
orthofinder将在dir1即输入数据目录下,新建以Results_开头后缀日期的文件夹,用于结果的存放,其中主要的结果为Orthogroups.txt。
示例如下:
OG0000000: GCA_000069225.1|ORF_00051 GCA_000069225.1|ORF_00125 GCA_000069225.1|ORF_00135 ...
OG0000001: GCA_000069225.1|ORF_00003 GCA_000069225.1|ORF_00028 ...
Orthogroups.txt的格式与OrthoMCL的计算结果的格式完全一致。每行以Orthogroup的ID开头,:后显示每个蛋白质的locus tag(用空格分隔)。
