Published at 2020-07-07 12:50
Author:zhixy
View:5182
马尔科夫聚类算法(Markov Cluster Algorithm,MCL)是一种快速且可扩展的无监督图聚类算法,常被应用于生物信息学领域 ,是一种简易有效、适应性强、可拓展的聚类算法。
MCL的主要思想是模拟图中的随机游走的过程。位于同一簇的点,内部联系紧密,而和外部联系较少,即任意选一点出发,它的一阶邻居节点和这点在同一簇的可能性远大于离开当前簇到达新簇的可能性。
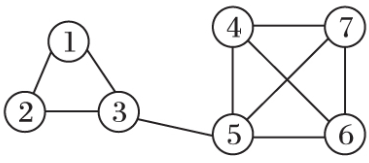
在上图所示的网络关系中,全部的7个节点直观的可见有两个簇和
,这两个簇只有一条边链接。这些单个节点明显的与簇内的节点关系更密切,而与簇外的节点关系疏远。MCL算法的目的就是要找出这种成簇的聚类关系。
假设每一条边都是一样的(均权重)。如果从节点1出发,到达节点2的概率是50%,到达节点3的概率是50%,到达节点4、5、6和7的概率为0。如果从节点2出发,到达节点1、3的概率是50%,到达其余节点的概率均为0。如果从节点3出发,到达节点1、2、5的概率均为33.33%,到达其余节点的概率均为0。通过计算可知每一个节点到达其他节点的概率,得到如下所示的概率矩阵。
如果我们遵循以上概率矩阵,在上一张关系网络中,随机的选择一个节点,开始游走,并且进行多次这样的“Random Walks”,那么就有很大可能发现簇群,达到聚类的目的。而“Random Walks”的实现则是通过马尔柯夫链(Markov Chains)。
马尔可夫链描述了一种状态序列,其每个状态值取决于前面的有限个状态。马尔可夫链是指具有马尔可夫性质的随机变量的数列。这些变量的范围,即它们所有可能取值的集合,被称为“状态空间”,而的值则是在
的状态,如果
对于过去状态的条件概率分布满足下式:
则称其是一条马尔可夫链。马尔可夫链模拟马尔可夫聚类算法中的随机游走过程,能够得到相应概率矩阵。马尔可夫聚类算法经历多次随机游走过程,可较大概率地发现簇群,最终得到聚类结果。
第一步,根据节点所代表的对象,以及对象间关系构建网络,并转换为添加自环(消除奇偶幂次导致结果无法收敛的影响)的邻接矩阵。
第二步,通过对邻接矩阵的标准化,得到概率矩阵
。
第三步,每次对矩阵进行次幂方,一般e值取2。该步称为Expansion操作,得到矩阵
。
第四步,对矩阵内的每一个值,进行
次幂方,然后将求出结果除以所在列中的
个元素的
次幂方的和,得矩阵
。主要作用是将概率矩阵中的每一个值进行
次幂次扩大化,使得紧密的点更紧密,松散的点更松散。该步称为Inflation操作。
第五步,标准化Expansion和Inflation操作后的矩阵,得到矩阵。
第六步,重复第3-5步,直至收敛。即,判断是否与Expansion操作前的矩阵近似相等,也就是矩阵中任意同一位置元素值相差小于0.01。如果两矩阵收敛至近似相等,则输出聚类结果。最终,结果矩阵变成聚类的聚簇。
基因家族分析的目的,即对所有受试基因组中所有基因,通过判断两两基因间的相似性,来将这些基因进行聚类,且通常认为聚类成簇的基因属于同一个基因家族。
目前主流的基因家族分析软件,如OrthoMCL和OrthoFinder,都是采用图聚类的方法,也就是利用了MCL。这些软件无一例外都是通过Blast首先建立基因/蛋白序列之间的关系,然后再转换为网络关系,近而利用mcl软件进行聚类的。
建议通过conda安装mcl。
(base) [user@server ~]# blastp -db allprotein.fasta -query allprotein.fasta -outfmt 6 -evalue 1e-5 -out all-to-all.out
(base) [user@server ~]# cut -f 1,2,11 all-to-all.out > seq.abc
通过cut命令,将blast结果中的第1(query id),2(hit id),11(e-value)列提取并保存到seq.abc文件中。
(base) [user@server ~]# mcxload -abc seq.abc -write-tab seq.dict -o seq.mci –-stream-mirror –-stream-neg-log10 -stream-tf 'ceil(200)'
–-stream-neg-log10对网络边的权重(e-value)取负对数转换,并通过-stream-tf 'ceil(200)'对结果进行封顶,即最大值到200。
(base) [user@server ~]# mcl seq.mci -I 1.5 -use-tab seq.dict -o mcl.I15.out
在聚类结果mcl.I15.out中,一行代表一个聚类组,组内每个成员的ID由tab分隔。
参数
-I即对Inflation操作中幂次扩大化时,值的设定。一般设定
-I 1.5,当该参数越小,聚类的组越少,过小时(如<1.0),可能导致mcl不会收敛。
mcl分析的过程相对比较简单,其中关键的参数只有一个。因此决定最终聚类结果优略的,主要是初始关系网络。在基因家族分析中,也就是all-to-all blast的结果,决定了最终的聚类。
Stijn van Dongen, Graph Clustering by Flow Simulation. PhD thesis, University of Utrecht, May 2000. (download)
Enright A.J., Van Dongen S., Ouzounis C.A. An efficient algorithm for large-scale detection of protein families. Nucleic Acids Research. 2002; 30(7):1575-1584. DOI: 10.1093/nar/30.7.1575
Stijn van Dongen, Cei Abreu-Goodger. Using MCL to Extract Clusters From Networks. Methods Mol Biol. 2012; 804:281-95. DOI:10.1007/978-1-61779-361-5_15
