Published at 2020-06-30 16:53
Author:zhixy
View:8888
Paschalia Kapli, Ziheng Yang & Maximilian J. Telford. Phylogenetic tree building in the genomic age. Nature Reviews Genetics. 2020 21:428–444. DOI: 10.1038/s41576-020-0233-0
物种是如何联系的是进化生物学的基础问题。了解物种关系不仅是进化生物学自身的重要目标,也支撑了系统发育分类系统。生命之树是研究新的表型起源和生物进化过程的基础框架。将可遗传的表型和基因型特征映射到一颗进化树上,是各种不同的进化分析的基础,这使得我们可以推测特征的同源性,也可洞悉关于特征的丢失和趋同进化。存在于两个分类单元共同祖先的特征被定义为同源特征,借此我们可以去了解他们的祖先的一些特性。特征映射让我们可以在进化树上追踪特征的变化,近而去构建进化的历史路径。进化树(和分子数据)也是将时间尺度拟合到进化过程的方法基础,同时也是用来建立进化过程趋势的方法基础。
重建所有生命之间的关系,虽然早在亚里士多德和林奈的分类尝试中就有了先例,但直到19世纪达尔文主义时期的才真正开始。虽然进化树最初在很大程度上基于形态特征,但生物分子-核酸和蛋白质-为重建进化树提供了更强大和丰富的信息来源。自从DNA测序和序列数据首次用于系统发育分析以来,我们对生命之树的理解发生了根本性的变化,并在达尔文的梦想方面取得了重大进展。
Darwin’s dream: "very fairly true genealogical trees of each great kingdom of nature".
近二十年来,分子系统发育依赖于一个或几个基因的数据,通常是通过PCR扩增和Sanger测序产生的。新的测序技术的发展导致了大量数据的产生。基因组和转录组测序的简易性和低成本也意味着可以考虑的分类群数量正在大量扩大,这在最近关于对地球上所有物种基因组进行测序的建议中得到了体现。重建生命之树的数据越来越多,但准确的树重建并不总是容易实现的。
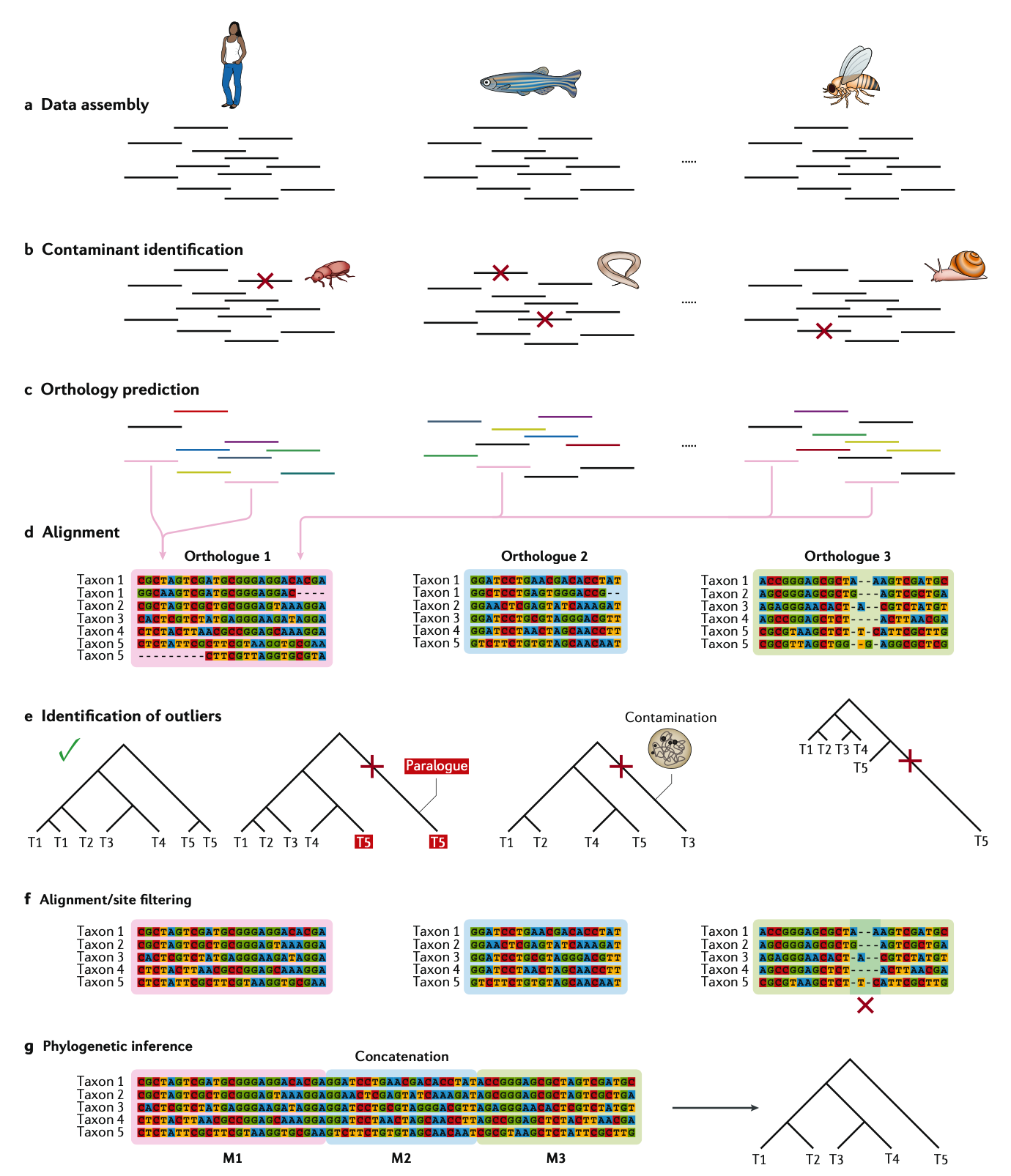
在这篇综述中,作者描述了系统发育分析中的主要步骤(上图,涉及数百个或数千个基因),即所谓的系统发育基因组学方法。对于每一步,作者都概述了各种方法的选择,以及模型复杂性和计算需求之间的权衡。首先从一组基因组或转录序列中鉴定同源基因(即其关系将可靠地反映物种关系的基因)开始。 然后,讨论如何比对来自不同物种的直系同源基因,以解释插入和删除,以及修剪不可靠比对区域的策略。 最后,详细讨论了推测方法和替代模型的选择,以及识别和避免或减轻潜在误差的方法。
分子系统发育分析开始主要是使用一组小的通用同源基因,包括核糖体RNA和(对于真核生物系统发育)线粒体基因组。 rRNA的广泛使用源于使用通用引物进行PCR扩增的难易程度(不像蛋白质编码基因,需要简并引物),这些通用基因之间的同源性是明确的,且有这些序列的数据库也比较健全。
近年来高通量测序技术的进步意味着基因序列数据在序列数据库中是丰富的,新的数据是廉价和容易产生的。 我们现在面临的数据收集的挑战是确保数据不受污染物的影响,识别能反映物种关系的同源基因,理想情况下,选择那些不太容易产生可能导致不准确进化树偏差的基因。
最初的基因序列数据可以来自基于基因组序列的基因预测(甚至来自草稿质量基因组),也可以来自mRNA的测序文库产生的转录本。 这一步骤的一个重要部分是识别和消除污染(无论是细菌、共生、寄生虫或肠道内容物,还是DNA提取后的交叉污染)。
两个基因,如果它们是从祖先基因遗传的,那么他们就是同源的。直系同源是一种特殊的同源性,来自不同物种的直系同源基因的分歧源自物种分化。所以,因此,同源基因描述了产生它们的物种之间的关系。其他同源性,包括旁系同源(源自基因复制时间)和外源同源(源自较远物种的横向基因转移)。旁系同源和外源同源不能反映物种的进化关系。所以,确定直系同源基因就成为了重建物种系统发育历史的关键步骤。在不同世系(Lineage)中,基因复制和丢失是较为常见的,这无疑增加了直系同源基因鉴定的难度,因为它们会导致旁系同源基因被错误鉴定为直系同源基因(即使是单拷贝基因)。
从头鉴定直系同源基因的方法可分为两大类:基于进化树的方法和基于图的方法。
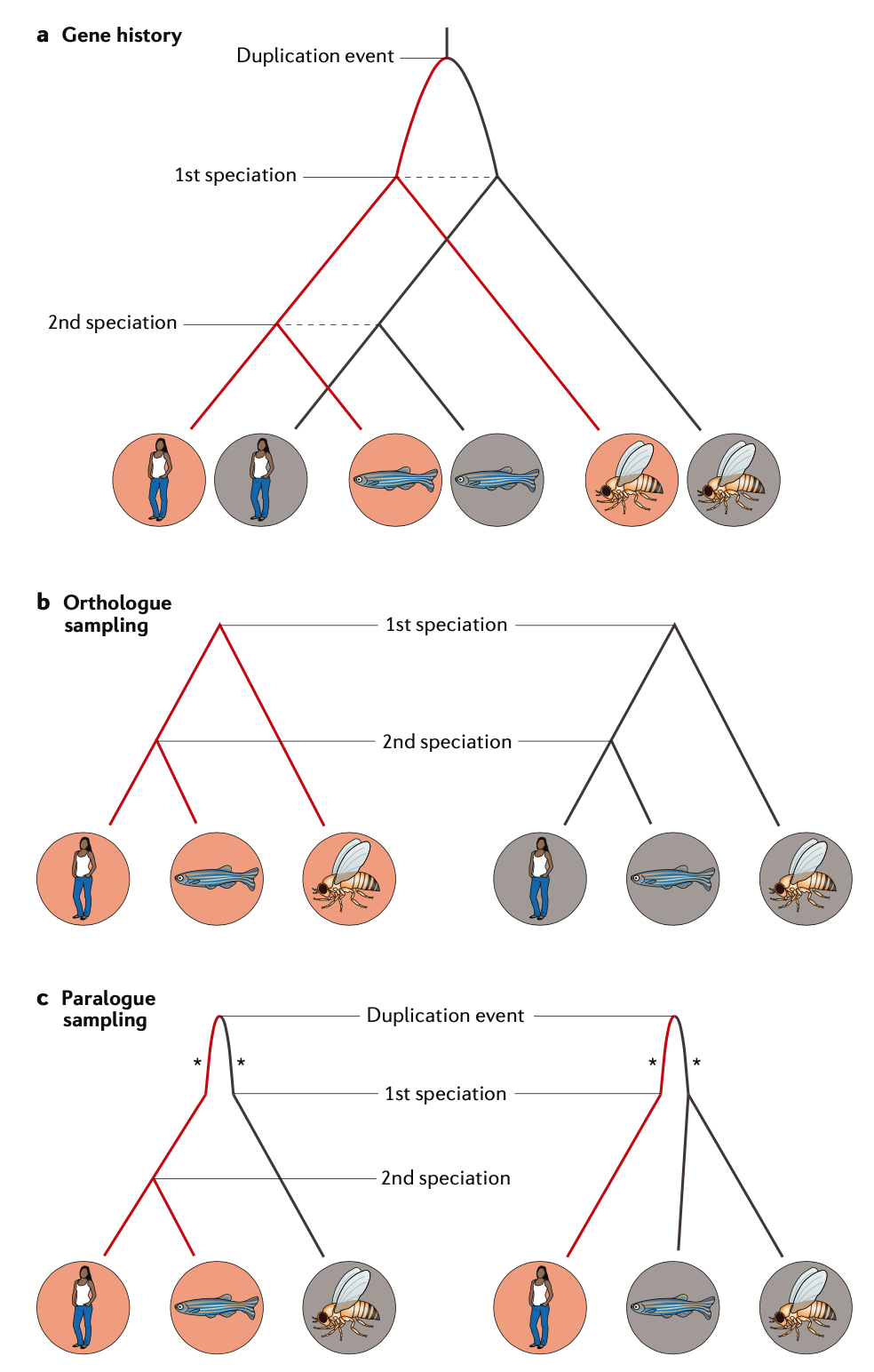
基于树的鉴定方法,通过比对同源基因并建树,然后找出那些可能与物种分化有关,而与基因复制或横向基因转移无关的同源基因。这些方法在概念上最接近直系同源的定义,但在计算上是昂贵的,因为它们需要整个基因家族的比对和系统发育推断,而且这些家族通常包括数百个序列。 序列间较深的分歧,对基因树的推断提出了更大的挑战,因为系统发育信号被侵蚀(即多个突变累积导致同质性),系统性错误发生的风险也会增加。 如果不考虑导致基因树不一致的其他过程,如不完全谱系分类、水平基因转移、杂交、导入和非等位基因转换,则基因家族关系可能会进一步模糊。特别是,在特定的生物群中,一些过程的频繁发生,例如频繁发生HGT的细菌,和发生杂交、基因组复制和多倍体化的植物,这使得它们更有可能遭受直系同源基因预测错误。
基于图的直系同源基因鉴定方法依赖于这样的假设,即:相较于其它同源基因,一个物种中的一个基因应该与第二个物种中它的直系同源基因更相似,反之亦然。这一定义导致了目前最流行的基于图的方法:双向最优匹配法和一些其他衍生方法。这些方法都是基于两两基因组的序列相似性的比较(BLAST)。基于图的方法并不能避免基于树的方法所涉及的问题,但它的优点是计算效率高,并且可以很好地与大型数据集进行结合。
鉴于序列和基因进化的复杂性,直系同源基因从头预测的方法必然是近似的。令人鼓舞的是,基于这些程序的系统发育研究产生了一致和准确的系统发育分析结果。然而,直系同源预测错误可能是导致系统发育分析中不一致结果的根源之一。一种替代方法是使用一组直系同源基因做参考,在新测序的物种中识别它们的共同同系物。几个专门的数据库提供适合于这一情形的直系同源序列,有些跨越生命的所有域,例如OrthoDB和OMA,还有一些侧重于植物(Plaza)和哺乳动物(OrthoMam)等特定生物群)。有几个管道工具可用于自动化此过程,并且遵循此策略有两个优点。首先,它在计算上成本更低,其次,它可以减轻与不完全基因抽样相关的错误。特别是当使用转录数据时,这一点特别明显,因为转录数据通常只包含一部分基因。数据的不完整性与差异基因丢失可能会增加从头预测中同系物的误识别。通过使用基于高质量基因组数据的直系同源基因参考,以保证研究目标基因库的完整性,可有效降低上述风险。
由于直系同源基因的鉴定和物种系统发育的推断是相关联的,所以直系同源的假说也可以在物种系统发育分析中进行检验。 特别是,多拷贝基因数据可以同时用于估计物种和基因家族进化。现在已经出现了几种方法,它们采用完整的贝叶斯框架或启发式方案,对这些方法性能的比较评估显示出有希望的积极结果。
直系同源的鉴定中最后一个需要考虑的问题是作为分析单位的遗传片段。使用基因(全部或部分)作为识别物种基因组同源部分的手段是典型的。然而,大多数基因由多个结构域组成,随着时间的推移,它们的顺序和数量可能会发生变化。 在这种情况下,有人认为结构域可能是更适合于直系同源的单位,因此可以用于系统发育推断。
由于插入和确实的存在,不同物种中的同源基因或蛋白一般都有不同的长度,并且即使有相同的长度,在同一个位置上的残基也可能不是同源的。识别跨基因的同源残基需要对齐基因,通过在序列中添加空位,以便在最终的多序列比对中,比对的每一列中的残基都应该来自相同的祖先残基。精确的对齐序列在进化关系的推断中最基本的环节,但是,对于indels经常出现的基因来说,这是一项具有挑战性的任务。 当对齐蛋白编码DNA序列时,核苷酸自然地以密码子为单位进化,而不是单个核苷酸。 这一特性,以及氨基酸序列变化速度低于相应核苷酸的事实,意味着在蛋白质水平进行的对齐通常要比在DNA水平上对齐要合适,然后根据氨基酸的对齐来指引DNA以密码子为单位对齐。
对齐方法可分为三大类。 最常用的方法是采用渐进的方法,包括Muscle、Clustal和MAFFT。 这些方法首先对每一对序列的相似性进行粗略估计,并利用这些信息生成序列之间关系的近似引导树。 然后,根据引导树,它们首先对最相似的一对序列进行对齐,并逐步将更远相关的序列添加到这种固定的对齐中,从而建立对齐。
其次是基于一致性的方法,包括T-Coffee、ProbCons和MAFFT的一些版本。 最初,这些方法估计所有成对对齐,对于每个序列对,保存一个替代高分解的记录。 随后,他们试图确定总体对齐,以最大限度地提高所有对之间的一致性。 基于一致性的方法比渐进方法慢,但总体上更准确。
最后,计算成本最高的是基于统计或进化的方法,如Bali-Phy和StatAlign。 这些假设是插入和删除的显式进化模型,并在贝叶斯框架中共同推断序列的对齐和树。统计方法是最合理的;然而,对于大型数据集,它可能会在计算上有很高的需求。 在这种情况下,使用良好的启发式方法,如PRANK和MAFFT,是一个合适的折衷方案。特别是对于更深分歧的序列,MAFFT(‘MAFFTE-INS-I’和‘MAFFTL-INS-I’)的版本,它们分别改变了长内部或终端间隙的可能性,可能是可用的替代方案。
任何直系同源基因鉴定程序都可能错误地将污染物、类似物或远源同系物识别为直系同源。 这些错误可能会影响系统发育推断的准确性,例如,产生较长的分枝、有偏差的模型参数,甚至改变树的拓扑结构。为了尽量减少这种误差来源,系统发育基因组分析项目遵循旨在识别离群序列的方法,通常采用基于BLAST的序列比较来测试最近的邻居与系统发育期望的兼容性。例如,一种真正的昆虫同系物有望显示出与来自双栖门的同系物比来自非双栖门的同系物更高的相似性,如果不满足这样的假设,则可以从中删除该序列。 这些方法可以有效地进行数据清洗,但它们通常需要一些关于所涉及的分类群的系统发育关系的知识。
有几种工具可以自动化这种基于BLAST的过程,或者使用替代方法进行离群序列检测(例如,Phylo-MCOA基于多个共惯性分析),也存在旨在识别和消除可能与系统错误或低系统发育信息相关的特征序列的工具。最后,为了丰富可能是由太严苛的直系同源预测(即导致许多假阴性)产生的直系同源基因,可能需要在更宽松的标准下使用基于参考的直系同源预测管道工具。
随着序列分歧程度的增加,对齐质量自然降低。由于对齐错误可能会影响后续的系统发育分析,因此需要过滤不明确的排列区域。过滤可以基于关于对齐质量的特殊标准,如空位程度和序列相似性,或者只保留对对齐参数变化具有鲁棒性的对齐位置。关于对齐修剪对下游系统发育分析质量的影响的报道各不相同,因此修剪应谨慎使用。
给定一组对齐和修剪的同源基因,有两种方法来推导物种树。 首先,每个基因排列可以独立分析,以提供对树的估计,然后可以将不同的树整合起来,以产生对物种树的估计。 这就是所谓的超树方法。 第二,排列的基因可以连接成一个超矩阵,并对其进行分析,以产生物种树的全局估计。 虽然我们讨论了跨基因谱系异质性的一致性文本中多个基因树的协调方法(以下),但超矩阵法是最常用的方法,也是本综述的主要重点。
系统发育重建方法分为两类:基于距离的重建方法和基于特征的重建方法。距离方法包括计算每一对物种之间的遗传距离(基于对齐序列的比较),并迭代地使用由此产生的距离矩阵构造一棵树。最流行的距离方法是邻居连接(NJ)算法。由于NJ并不再在所有可能的巨大树空间中搜索(根据一定的标准)最优树,因此在计算上是非常有效的。有几种NJ方法或变体能够对数千个样本进行系统发育分析。然而,距离方法对于远距离的物种往往表现不佳,因为很难估计大的距离,而距离方法在定义成对距离时,通过求和物种之间分支长度的策略实际上加剧了这一问题。
基于特征的方法包括最大简约(MP)、最大似然(ML)和贝叶斯推理(BI)。最大简约法是在可能的树空间中搜索最优树,判定标准是这颗树在解释数据时,所需的核苷酸或氨基酸变化数量最小。这颗变化数量最小的树拓扑又被称为最简约树。对于大型数据集,不可能对所有可能的树进行详尽的比较(对于10个物种,有8.2×1021个可能的根树),所以通常使用各种启发式树搜索方法。简约法之所以有吸引力,是因为它的数学简单性和计算效率。然而,简约法显然进化过程的假设是不切实际的,更缺少一个显性的模型假设。该方法缺乏明确说明的模型,因此很难概括序列进化过程中众所周知的特征,例如特征状态之间的不同速率(不同的颠换和转换速率)和位点之间的不同速率(第三密码子位置的速率高于第一和第二位置)。同时,简约法比似然法更容易产生系统误差,包括长枝吸引(long-branch attraction, LBA)。然而,该方法对于那些难以设计出适当的特征进化模型的数据类型是有用的,例如基于基因组重排或独特形态特征的罕见进化事件。
与简约相比,ML和BI方法都是基于显式声明的序列演化模型和似然函数。在由未知参数θ参数化的统计模型下,似然值L(θ)是观测数据作为θ函数的概率。在这里,θ可以包括替换模型的参数和树上的分支长度。在系统发育学中,几乎所有的模型都假设对齐中的不同位置或列是独立的;似然值是在不同位置观察数据的概率的乘积。 似然函数包含模型下关于未知参数的数据中的所有信息。换句话说,使观测数据看起来很可能发生的参数值比使数据看起来几乎不可能的参数值更接近真实情况。因此,似然法就是计算让似然值取最大值时的参数值。最早,似然法是由Felsenstein引入了树估计的,现在已在PAML、PhyML、RAxML-NG、IQ-Tree和FastTree等程序中实现了树估计的ML方法。对于每个树拓扑,替代参数和分支长度进行优化,使似然最大化,达到最大似然的树拓扑即是ML树。
贝叶斯方法也依赖于显式声明的模型和似然函数。它不同于ML,因为它使用统计分布来量化参数中的不确定性。在观察数据之前,使用先验分布来描述我们关于物种树和模型参数的先验信息。在收集和分析数据后,后验分布也做了同样的事情。后验是先验乘以似然,重新标度,使其成为适当的分布。因此,后验从数据中捕获与参数相关的所有信息,并且是先验的更新。
贝叶斯方法在20世纪90年代被引入分子系统发育,并在MrBayes、RevBayes、BEAST1、BEAST2和PhyloBayes等程序中得到了实现。贝叶斯系统发育的计算是利用马尔可夫链蒙特卡罗(MCMC)算法实现的,它是一种计算机模拟算法,它从树的后验中生成树的拓扑和参数样本。在实际应用中,算法访问给定树拓扑的频率是对该树的后验概率的估计。最大后验概率树(或MAP树)是我们对真树的最佳估计。 95%可信的树集包括最可能的树,总后验概率≥95%;可信集有这样的解释,即给定数据和模型,该集有95%的概率包含真实树。
基于似然的方法(包括ML和BI)的一个严重缺点是它们的大量计算需求,因为它们可能需要数千个CPU小时才能运行;MCMC算法尤其如此。似然函数的制定需要明确说明关于序列演化的模型假设;有些人认为这是一个缺点(因为所有的模型都是错误的)。但是,假设的模型可以被测试,它对分析的影响可以被评估,并且可以通过结合进化过程的重要特征来改进模型。事实上,统计系统发育的大多数现代发展都是在似然框架下实现的。
我们可以认为NJ树、MP树或ML树是对真实系统发育树的点估计。在点估计中附加置信度是可取的,就像常规参数上的置信区间那样。最常用的方法是自展法(Bootstrapping),也是由Felsenstein引入的。该方法将产生多个(比如100),从原始数据中重新采样和替换而得的伪重复数据。然后对伪数据集进行与原始数据集相同的分析。树的Bootstrapping支持是在伪数据集中推断树的频率。Bootstrap通常用于表示对进化分枝的支持度(而不是整个树),对分枝的支持是在基于Bootstrap数据集的系统发育树重建之后出现同样分枝的频率。与其他统计应用中的Bootstrap不同,系统发育Bootstrap没有得到很好的接受,简明直接的解释。
应用Bootstrap法可以对距离树、MP树和ML树进行置信度评估。而对于贝叶斯法,树和分枝的后验概率提供了信心的自然度量,因此Bootstrap是不必要的。
在系统发育基因组的分析中,一个常见的现象是Bootstrap和后验支持值非常高(接近100%),无论关系是否正确。这对于贝叶斯后验概率特别明显。在系统发育基因组尺度数据集中,随机误差变得不重要,这种对不正确关系的强烈支持通常来自系统误差。
系统发育推断的误差主要有两种。随机误差是由于数据集的大小有限(即有限的对齐位点),而系统误差是由于违反了方法中的模型假设。 一般来说,当系统发育是在一个简单的同质过程序列进化模型下推断的(假设特征状态之间、位点或基因之间以及跨分类群或时间之间的同质进化速率)时,就会产生系统误差,因为现实中,这一过程往往是异质的。近年来序列数据的爆炸性积累意味着系统发育分析中的随机误差大大减少,但系统误差实际上随着比对序列的延长而增加。
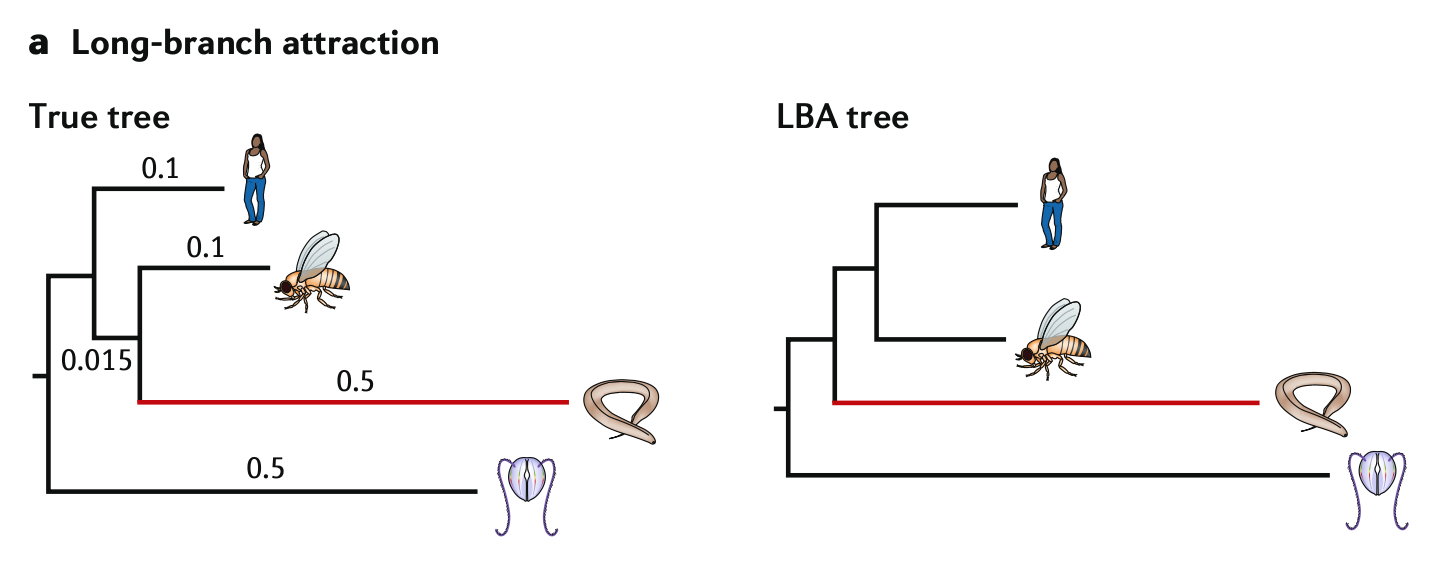
长枝吸引LBA可能是影响系统发育重建的系统误差中最著名的。LBA的根源是不同谱系中不平等的进化速率;导致每个谱系预期变化量的偏差,预期变化量由树上的长枝(高度分歧序列)和短枝(较少分歧序列)表示。LBA表现为树上长的,但实际上是远相关的分支的不正确聚类。两个不相关的长支,由于都有较多的替代,所以偶尔会经历相同的替换。简约的方法将重建这些趋同性,作为从一个共同的祖先继承下来的同源共享特征。似然方法(ML和BI)对LBA误差的鲁棒性比简约性强,因为它们是分支长度感知的,因此考虑了在两个长分支上收敛的可能性增加。然而,如果假设的替代模型是不正确的或过于简单的,例如错误地假设站点之间的均匀变化率,ML和BI可能会受到LBA的影响。
在实际数据集中,LBA可能很难识别。它的症状包括两个或两个以上快速进化的世系组合在一起或一个长枝分类群连接到一个遥远的外群。所以,重要的是评估这种关系对替代模型变化的鲁棒性。
已有几种特定策略来减轻潜在的LBA,例如排除进化速率很高的、有问题的物种,去除速率很高的基因或基因区域(这也往往具有较差的对齐质量),以及添加有助于打破树上长分枝的物种。最近,分枝长度异质性的测量被用来识别出现较少的速率异质性的基因,因此被认为对LBA不太敏感。本着同样的精神,有方法从单个基因树中识别和去除大量较长的分枝,从而减少速率异质性。
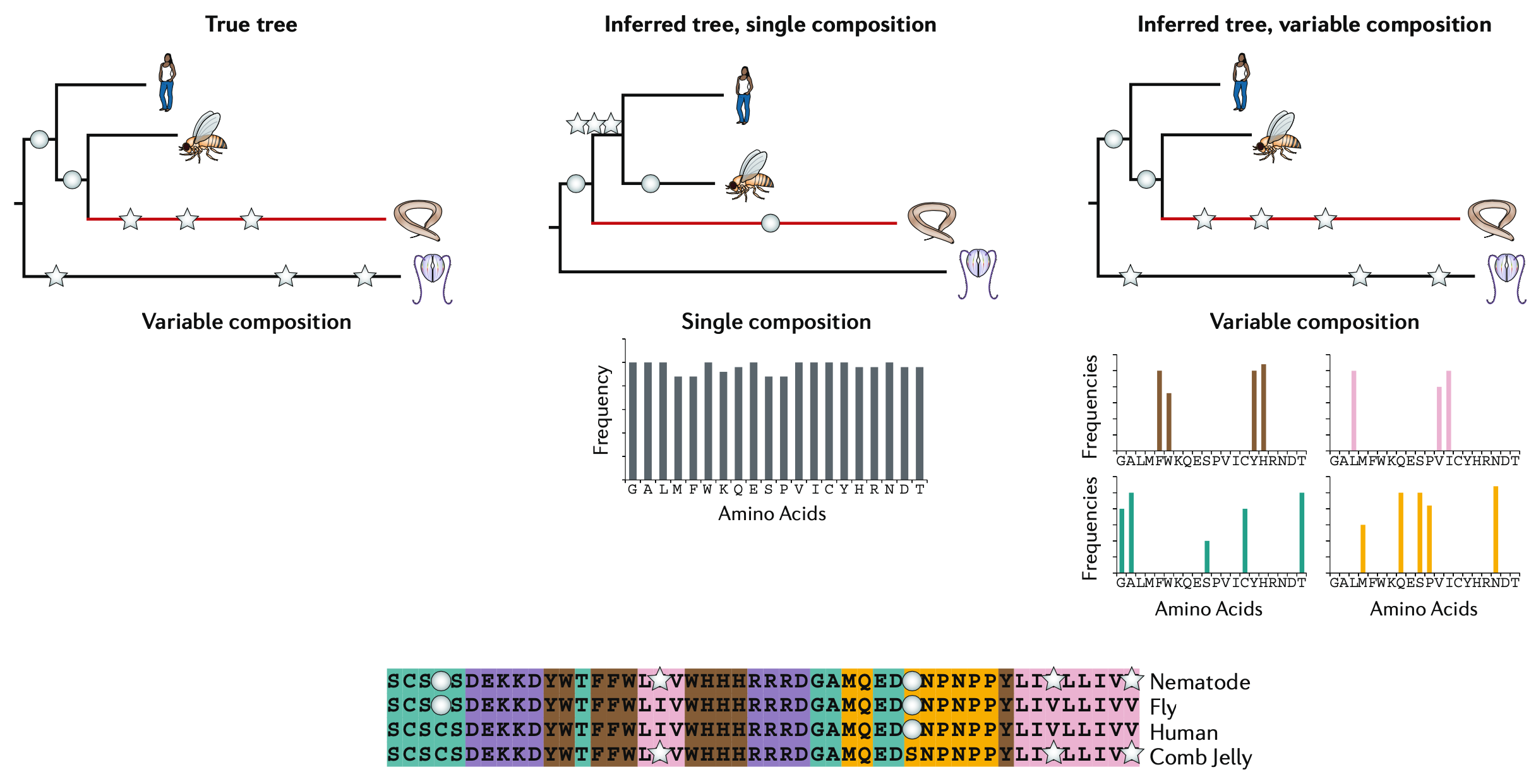
大多数系统发育推断模型假设在所研究的物种的整个历史过程中,替代过程是固定的,因此所有物种的4个核苷酸或20个氨基酸的频率是一样的。这种组分同质性的假设在对远缘物种的分析中经常被违反;一个明显的例子是,远缘相关的分类群已经独立地进化出富AT的基因组。在这种情况下,同质模型的假设将倾向于将具有相似碱基组成的物种聚类在一起,而实际上他们是远缘的。
处理组分频率异质的最佳方法是放宽对组分同质性的假设,允许特征状态频率参数在系统发育过程中漂变。这种模型涉及到树上每个分枝的一组频率参数,这将导致参数的大量增加,计算成本很高。
一个更实际的方法来避免这个问题是识别和删除有组分频率偏差的基因和分类群。有几种评估组分频率偏差的方法是可行的(例如,在软件包p4、IQ-Tree和PhyloBayes)。然而,如果最有偏差的分类群是我们的研究目标,或者如果大多数基因未能通过同质性测试,则不可能去除基因或分类群。
最后提出的方法是汇总字符状态。 例如,4个核苷酸可以被重新编码成嘌呤(A和G)和嘧啶(C和T),从而消除任何AT偏差。同样,20个氨基酸被重新编码成一个约简集,根据它们在替代矩阵中所表示的相互交换性分组。 重新编码自然导致信息丢失,这本身可能导致拓扑变化。然而,当数据被重新编码时,检查组分频率不同的分类群的位置是如何变化的,这可以提供有用信息。
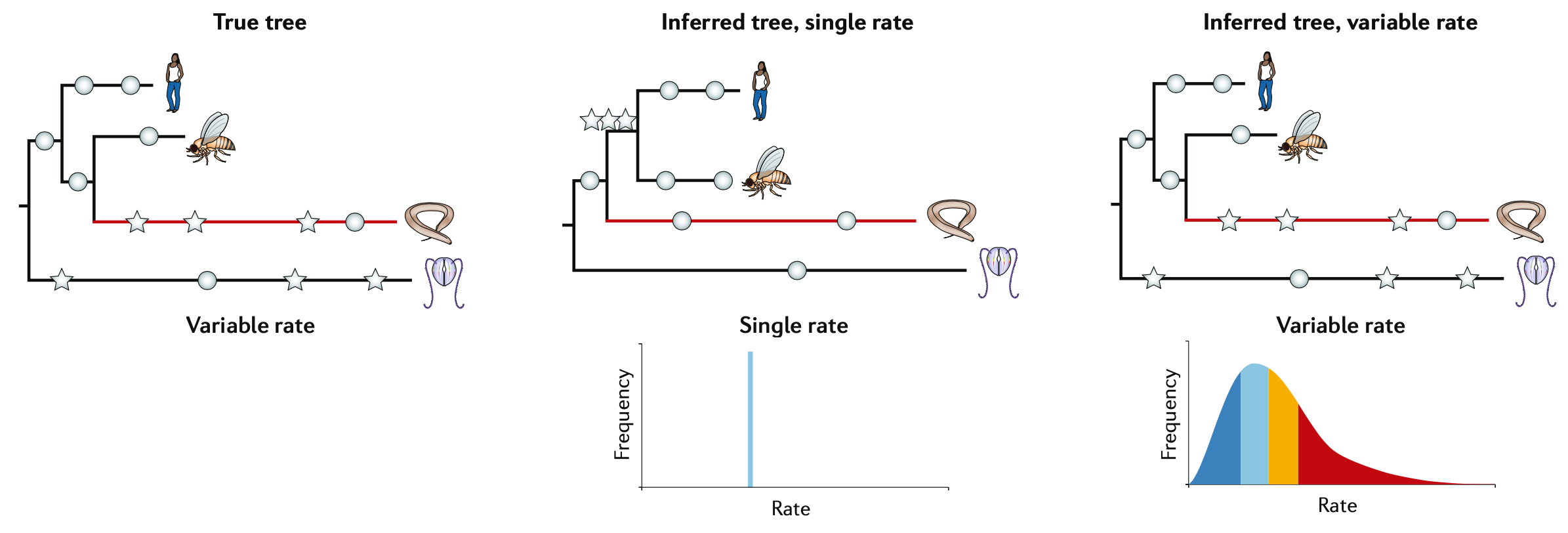
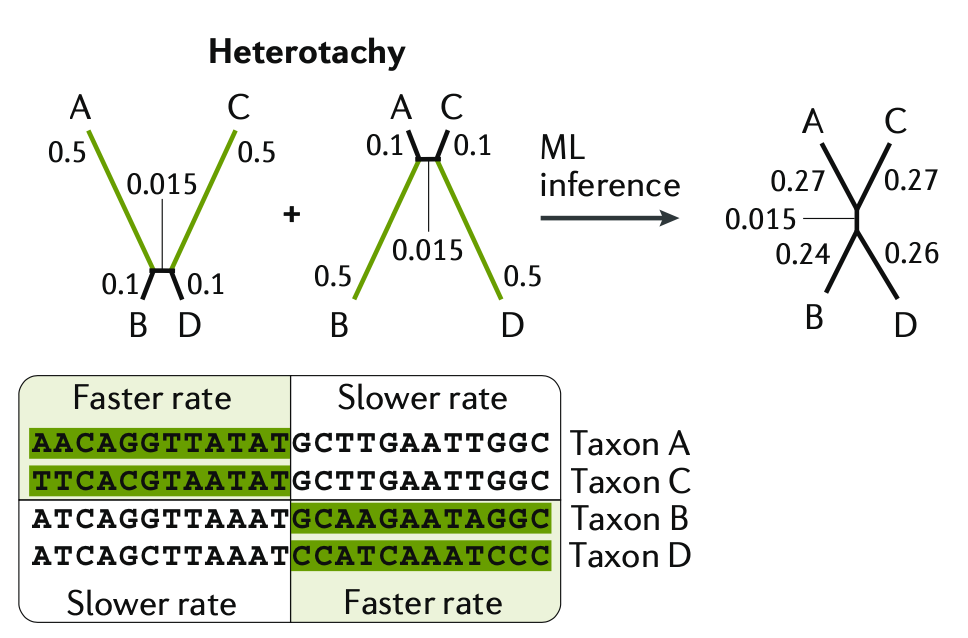
基因组的不同部分会以不同的速率进化。胶原的变化比组蛋白更快,内含子的变化比外显子更快,密码子中的第三个位的变化比第一和第二个位更快,蛋白质中的一些氨基酸处于强稳定的选择下,而另一些氨基酸则可以自由变化;最终,假设基因位点之间的恒定速率是不现实的。 假设一个单一(平均)速率导致系统低估了在具有较高速率的站点上发生变化的可能性。正如我们所看到的,低估变化的可能性(因此也是收敛演化的概率)往往会加剧LBA。为了适应这种位点间的速率变化,Yang提出了一个基于伽马分布的随机变量的来为位点速率差异建模。所得到的模型由后缀“Γ”或“G”表示,可以与任何核苷酸或氨基酸替代模型(例如,JCΓ、GTRΓ或LGΓ)相结合。在所有系统发育推断和模型选择工具中,都实现了计算位点间速率异质性的策略。适应位点间速率变化的替代模型包括自由速率模型(它假设几个离散的速率类)和伽马混合模型(它假设两个伽马分布的混合)。除了对齐序列中不同位点的异质性外,替代率和过程也会随着时间的推移而变化,这可能反映了不同分类群中蛋白质的结构和功能变化。因此,在一个特定的位点上的替换率和模式可能在系统发育的谱系之间有很大的差异;这种现象被称为“heterotachy”,目前处理它的方法仅在计算上,对非常小的数据集上进行树搜索,或对较大的数据集进行单个树的比较,是可行的。
在系统发育中使用的马尔可夫模型中,考虑不同类型替换的不同速率很容易被实现。 例如,颠换和转换可以分配不同的速率,使用两个参数。一般时间可逆模型GTR假设所有核苷酸有不同的频率(即三个自由模型参数),以不同的速率(即六个交换性参数)相互替代。
对于20个氨基酸,一般的时间可逆模型将涉及209个参数(19个频率参数和190个替代参数)。该模型参数丰富,可以拟合中等大小的数据集。然而,在树搜索过程中估计这么多参数在计算上是高成本的。因此,从数百或数千个蛋白质序列分析中得出的经验氨基酸模型更多地被使用,包括Dayhoff、JTT、WAG和LG。还有经验模型是根据蛋白质的特定子集(例如病毒、叶绿体和线粒体)计算的;不同的基因将最适合不同的模型。
系统发育基因组学研究中的常见做法是将所有基因连接成一个超级基因,从而推断出一棵树。然而,基因在进化的速度和过程中可能有所不同。这种基因之间的差异可以通过分区模型来解决,分区模型为对齐序列构造了不同的分区,并使同一分区中的位点共享进化特征和模型参数,而不同的分区具有不同的参数。分区模型通过考虑速率和替代模式的大规模异质性,提供了一种减少模型误差的方法。
在一个由数百个基因组成的数据集中,有几十个模型可供选择,将模型分配给基因或构建一个分区策略并不简单。自动模型选择方法通常假设一个固定的树拓扑,并试图通过改变每个基因的替换模型来最大化数据的似然值。一些工具将模型选择的过程与分区方案的评估相结合,在这种情况下,适合同一模型的基因被合并成一个更大的分区。对于大型数据集,分区选择和模型优化的组合任务在计算上也是高成本的。在不同的替代方案下,使用经验数据的系统发育推断可能会导致拓扑结构、分支长度和统计支持的差异。模拟分析表明,优化的分区方案与基于生物常识的分区方案相似,而且这两种方法都比未分区的数据要好得多。
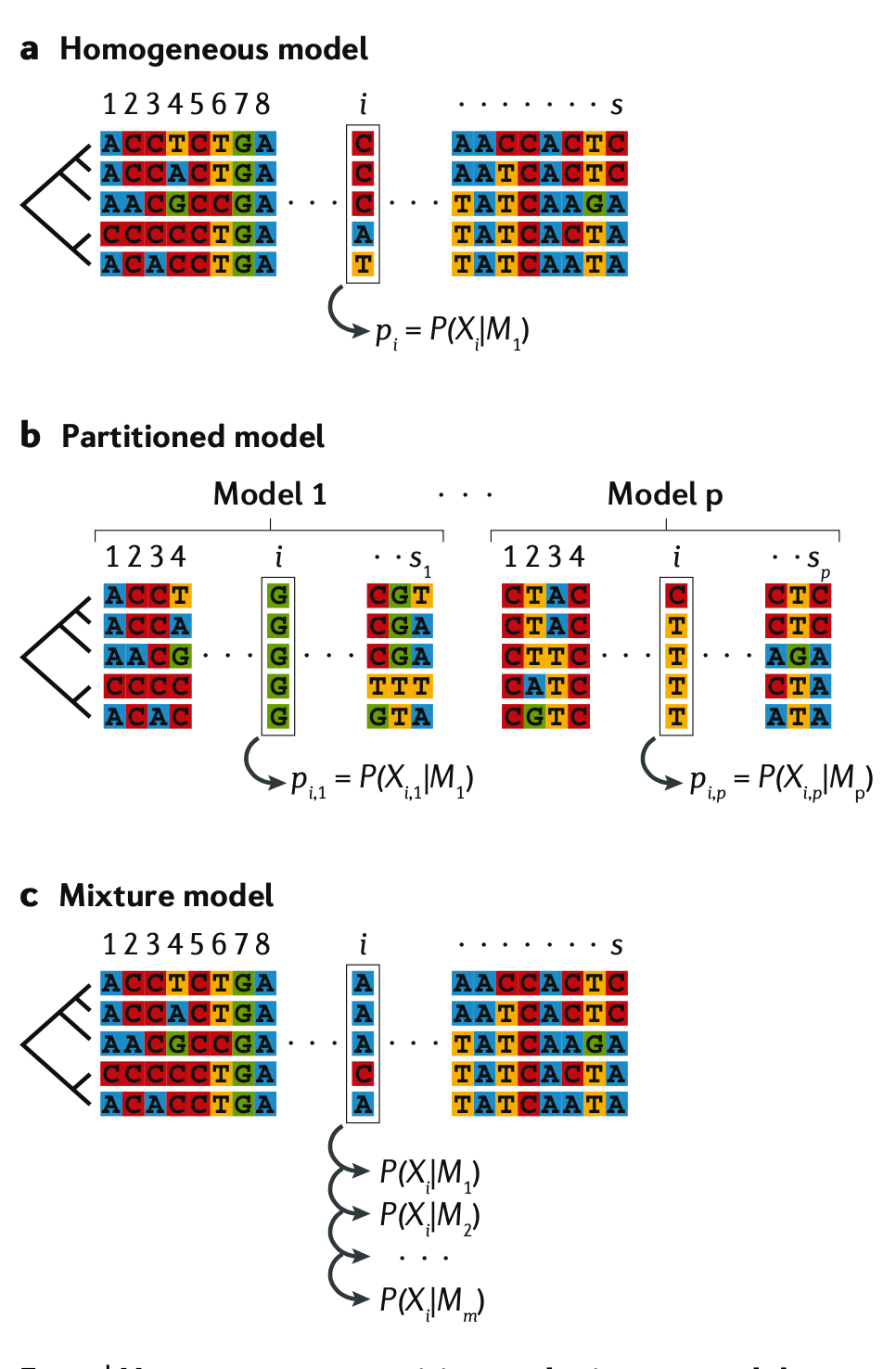
混合模型还可以适应替代速率和模式的位点间异质性。混合模型不将每个位点分配给特定的分区,而是将位点的所有可能分配平均分配给位点类。以上讨论的位点间速率的伽马模型就是一个典型的混合模型。当生物知识可以将位点分配给定义良好的分区(例如,将基因的位点分配给三个密码子位置)时,使用分区模型是很自然的;当缺乏这种知识时,混合模型提供了一种灵活的选择。
在蛋白质数据分析中,蛋白质的不同部分可能有非常不同的替代速率,以及对不同氨基酸的偏好决定了局部选择性约束。一刀切的经验替代矩阵,甚至是分区方法,不太可能在进化过程中捕捉到这些微妙之处。然而,混合模型可以自然地适应氨基酸替代率和位点替代模式的异质性。混合模型比分区模型涉及更多的计算,因为如果不知道每个站点来自哪个组别,就必须在似然计算中平均整个组。混合模型可以用来解释替代速率和模式中的位点异质性。该模型可以假设多个替代矩阵或多组氨基酸频率。Profile混合模型使用不同频率的20个氨基酸的多个组分,同时假设它们之间的一组交换率。 例如,C10-C60经验模型包括从已知序列的经验估计氨基酸频率。这些模型都是在贝叶斯和ML框架中实现的。在Phylo-Bayes中实现的“CAT”模型是Profile模型的最广泛推广。该模型将混合组分作为自由参数,并从数据中估计氨基酸频率和混合比例。重要的是,CAT模型和其他混合模型似乎不太容易低估分支长度,并且在对远缘物种的分析中比场地均匀模型更健壮。
将所有基因连接成一个超矩阵并推断出一棵树,假定了一个单一的基因树构成了所有基因的基础,并且它对应于物种树。然而,由于多种生物学过程,如祖先物种的多态性、基因复制和丢失以及水平基因转移-不同的基因或蛋白质可能有不同的历史或基因树。祖先多态性是指当我们及时追溯它们的历史时,来自不同物种的同源基因可能不会在它们到达共同祖先物种时立即汇合;因此,这些基因可能不会跟踪物种的系统发育,并且可能具有与物种树不同的树拓扑结构。这种现象被称为不完全谱系分选(incomplete lineage sorting, ILS)、深聚结(coalescence)或基因树-物种树不一致。如果物种树的内部分支较短,如果祖先物种的种群规模较大,则更有可能发生不一致。 即使分析方法忽略了ILS,以物种树中较长的内部分支为代表的系统发育关系也很可能被自信地解决。然而,对于通过辐射式物种发生形式产生的物种(在物种树中产生短的内部分支),ILS可能对物种树估计提出严重挑战。
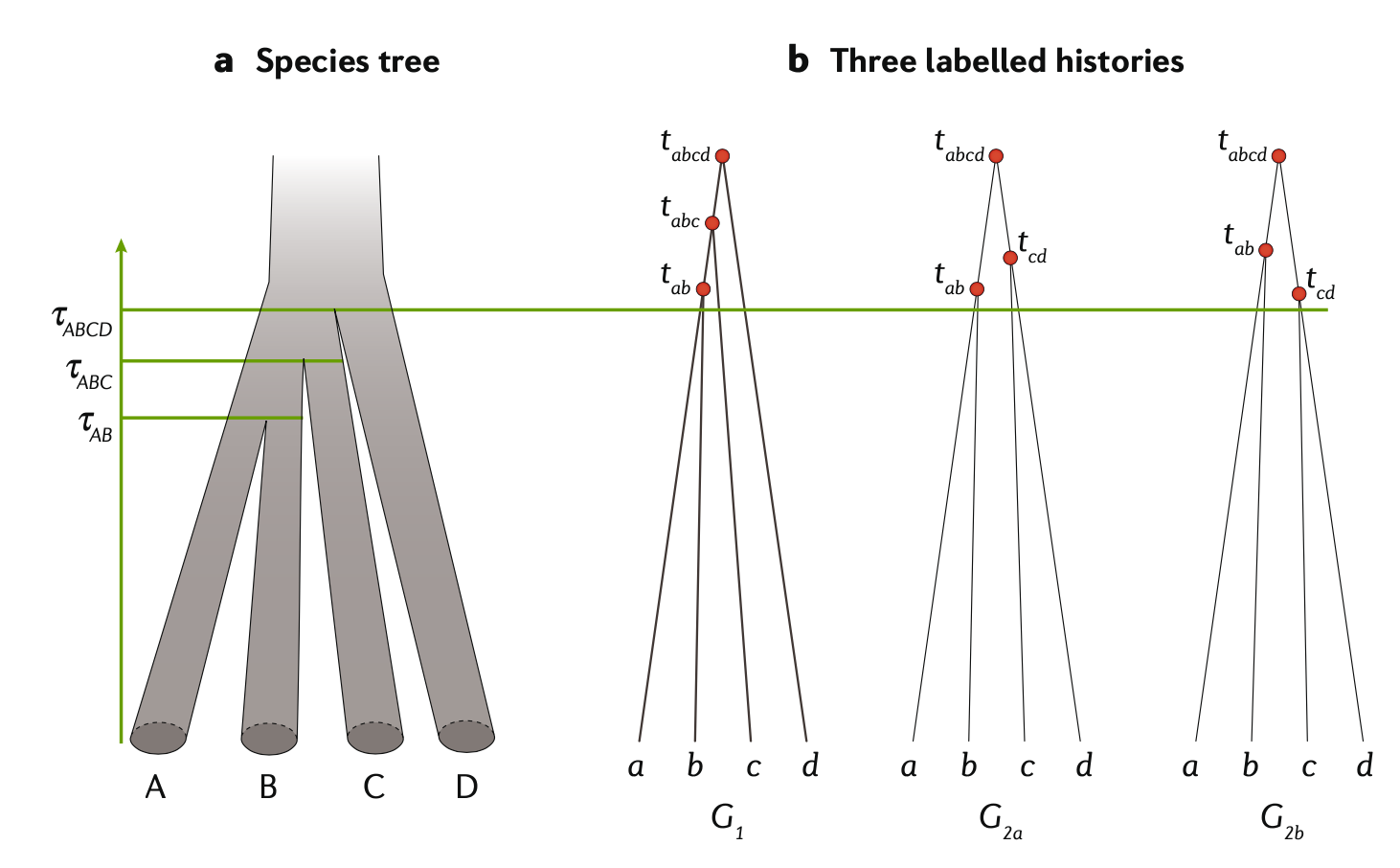
容纳ILS的框架是多物种聚结(MSC)模型,这是单种群聚结的扩展,适用于多物种的情况。 在MSC模型下,由于祖先物种的聚结过程,基因树(拓扑和分支长度)在基因或基因组区域之间存在差异:它们具有由物种树和物种分歧时间和种群大小等参数决定的统计分布。因此,MSC过程是生殖和遗传漂移的自然结果。简单的MSC模型已被扩展到包含跨物种基因流,导致了具有迁移的MSC(隔离-迁移或IM模型)和具有导入的MSC(MSCI或多物种网络聚结或MSNC模型)等模型。
有两种主要的物种树方法结合MSC模型。两步法使用系统发育程序推断单个位点的基因树,然后使用估计的基因树作为数据来构建物种树。流行的两步法程序包括ASTRAL和MP-EST。这些方法在计算上是有效的,可以分析数千个基因,但在重建的基因树中可能存在误差。
相反,全似然法计算对齐序列的似然值,从而适应基因树中的不确定性。实现MSC模型的常用程序包括BEAST和BPP;两者都是MCMC算法,涉及大量计算,尽管算法的改进使分析10,000个位点的数据集成为可能。
对模拟和经验数据的分析表明,全似然方法优于近似聚结方法和级联方法。许多基于聚结的方法在相对较浅的分歧中得到了应用和评价,但这些方法在重建深层系统发育方面的有效性尚不清楚。然而,ILS的根本原因是物种树中的短内部分支,而不是节点的浅度:深系统发育与浅系统发育一样受到ILS的影响。我们预计,今后几年将在评估和克服生命之树深层部分ILS的影响方面作出大量努力。
