Published at 2020-05-04 16:02
Author:zhixy
View:3676
基因组测序短序列,经组装软件如spades,velvet等组装拼接后,有必要对拼接结果进行评估。 目前最常用的评估软件,即QUAST。
输入基因组fasta文件,QUAST可直接统计fasta文件中的序列长度、GC含量、N50等指标,提供组装结果的基本信息。 在评估时如提供参考基因组,除了统计基本指标外,还会将组装结果与参考基因组序列进行比较,包括长度、GC含量、对齐程度等。 此外,除了单物种基因组外,QUAST还适用于评估宏基因组的组装结果(metaQuast)。
虽然QUAST官方网站提供了在线版本,但更常用的还是线下单机版本,推荐安装方式:
(base) [user@server ~]# conda install -c bioconda quast
(py27) [user@server ~]# quast -h
QUAST: Quality Assessment Tool for Genome Assemblies
Version: 5.0.2
Usage: python /opt/miniconda3/envs/py27/bin/quast [options] <files_with_contigs>
Options:
-o --output-dir <dirname> Directory to store all result files [default: quast_results/results_<datetime>]
-r <filename> Reference genome file
-g --features [type:]<filename> File with genomic feature coordinates in the reference (GFF, BED, NCBI or TXT)
Optional 'type' can be specified for extracting only a specific feature type from GFF
-m --min-contig <int> Lower threshold for contig length [default: 500]
-t --threads <int> Maximum number of threads [default: 25% of CPUs]
... (此处省略96行)
Online QUAST manual is available at http://quast.sf.net/manual
QUAST参数有很多,但通常我们使用QUAST主要用于基因组组装结果的评估,因此使用默认参数即可,无需设置高级的参数。
以下实例,以同一基因组(ERR182500)测序结果,
在设置不同Kmer值时,velvet的拼接结果为对象,利用QUAST比较和评估结果。
velvet的拼接Kmer分别设置为33,43,53,63, 73。
(base) [user@server ~]$ quast -o quast_out -t 8 k33.fa k43.fa k53.fa k63.fa k73.fa
在quast_out文件夹中,用浏览器打开report.html文件,查看结果:
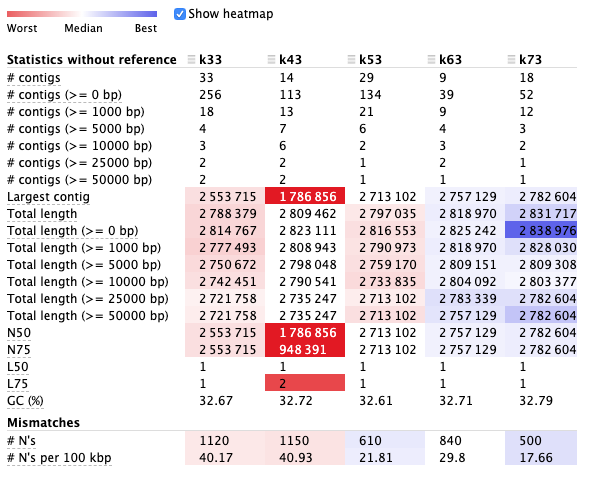
以上结果显示,Kmer=73时,QUAST认为拼接结果最好。
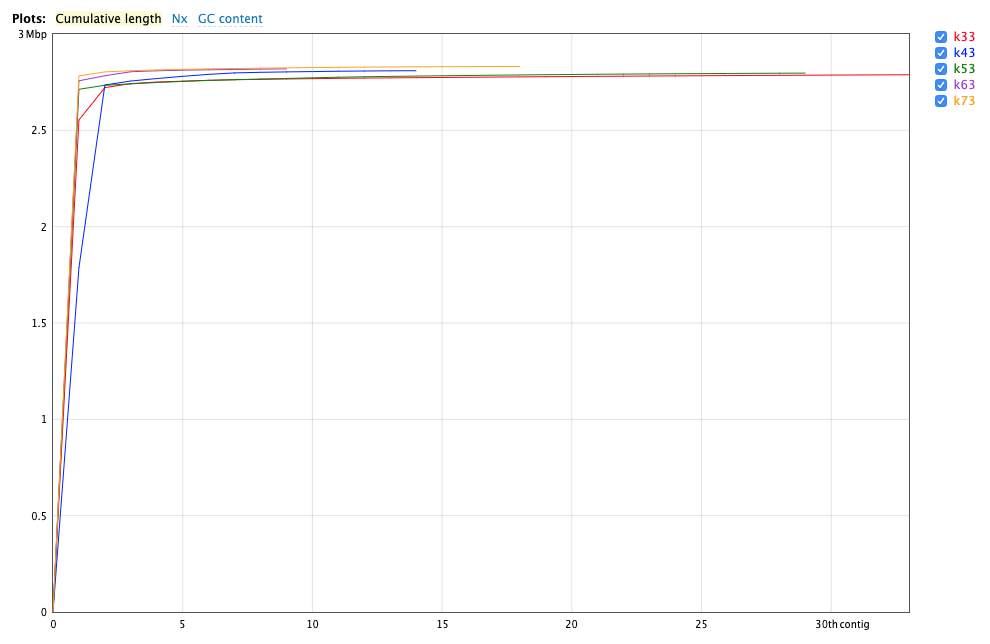
累计曲线显示相同的结果,即曲线越接近y轴,拼接效果越好。
Gurevich A, Saveliev V, Vyahhi N, Tesler G. QUAST: quality assessment tool for genome assemblies. Bioinformatics. 2013 Apr 15;29(8):1072-5. DOI:10.1093/bioinformatics/btt086
