Published at 2022-06-30 14:54
Author:lipp
View:3370
假基因是基因组中与特定蛋白质编码基因具有相似性,但由于存在移码、过早终止密码子或其他有害突变而无法产生功能性蛋白质的基因残体。
大部分假基因在染色体上都位于正常基因的附近,但也有位置在不同的染色体上的情况。假基因和正常基因结构上的差异包括在不同部位上的、程度不等的缺失或插入、在内含子和外显子邻接区中的顺序变化、在5'端启动区域的缺陷等。这些变化往往使假基因不能转录并形成正常的(mRNA)从而不能表达。
在此期间可能会发生 DNA 中的许多修改,例如插入、删除和移码。 这些修饰可能导致基因功能丧失并产生“未加工”或“重复”假基因。 有时,生态位损失或无效等位基因也可能导致一小部分“未加工”假基因。
即mRNA转录本经过反转录为cDNA,再插入基因组,由于插入位点不合适或序列发生变化而导致失去功能。
在种群或进化研究中,未被识别的假基因而不是真正的功能基因,可能会被错误放大。由于这些非功能性假基因不受选择压力的影响,它们的突变率可能异常高,因此利用包含未知的假基因极有可能导致错误的树。此外,假基因并不是垃圾DNA,它经历了进化事件,被视为基因组化石,默默记录着进化史。假基因还具有潜在的再激活能力。在某些时候,它们可以复活并在新陈代谢中发挥功能作用。因此鉴定假基因是理解作用于原核生物基因组的进化力量和编码在原核生物基因组内的功能潜力的关键步骤。
Pseudofinder是一种生物信息学工具,可从细菌和古菌基因组的注释基因库中检测假基因。借助 Pseudofinder 的多管齐下、基于参考的方法,我们可以识别高度降解且通常被基因调用管道遗漏的假基因,以及新形成的假基因,这些假基因可能只有一个或 一些失活的突变。 此外,Pseudofinder 可以检测到经历 relaxed selection的完整基因,这可能表明假基因的形成初期。 在注释管道中实施 Pseudofinder 不仅将阐明测序微生物的功能潜力,而且还将产生关于细菌和古细菌基因组进化动力学的新见解和假设。
假基因鉴定由基于参考的方法指导,其中感兴趣的基因组通过与用户提供的蛋白质序列数据库(例如 RefSeq [Pruitt et al., 2007])和/或密切相关的参考基因组。 使用蛋白质参考数据库,Pseudofinder 对截断、片段化和高度降解的基因进行基于证据的注释。 当有合适进化距离的参考基因组可用时,Pseudofinder 有能力检测隐秘的假基因,并报告失活突变的类型和数量(例如无义突变、移码诱导插入缺失等)。
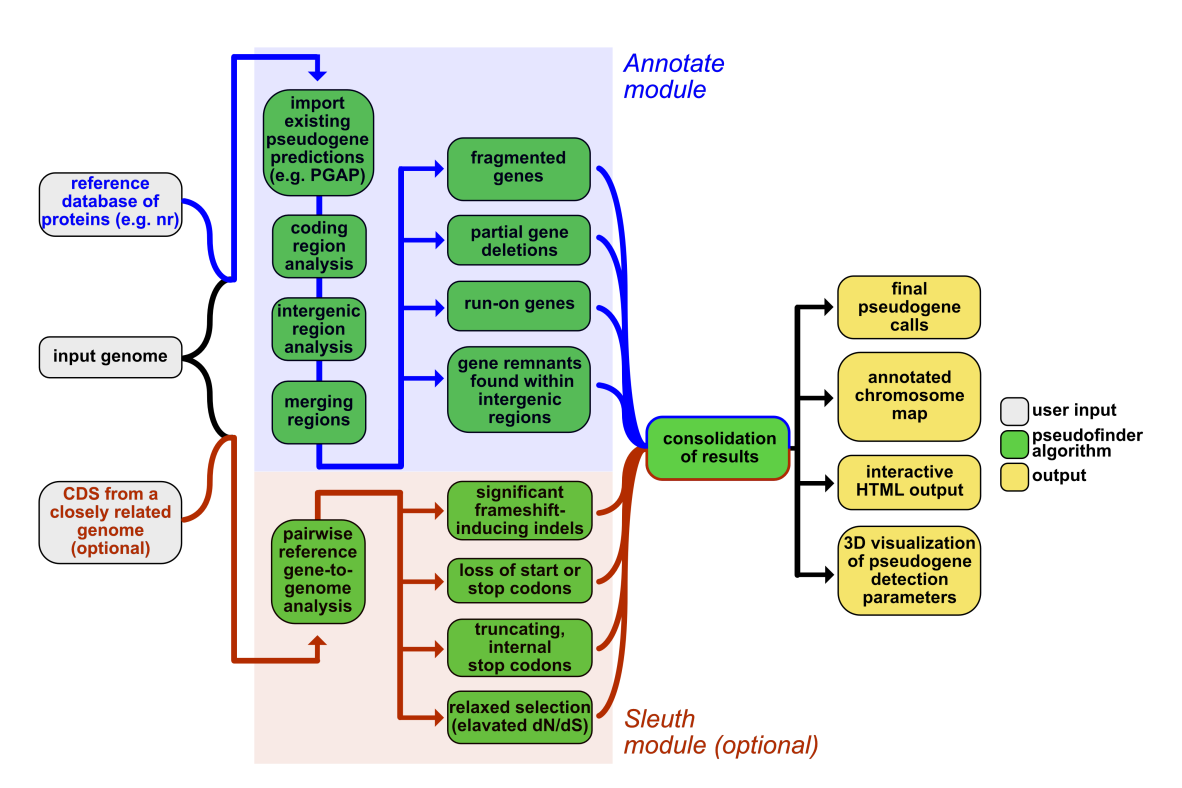
Figure 1: Pseudofinder workflow
Pseudofinder要求用户以Genbank格式提供基因组,以及为BlastP / BlastX搜索格式化的非冗余蛋白质数据库。如果可能,提供参考基因组允许Pseudofinder包括dN / dS计算以识别假基因。
数据库选择对于Pseudofinder的速度和灵敏度至关重要。用户可以提供他们想要的任何数据库,前提它是一个非冗余蛋白质数据库,用于BlastP / BlastX搜索。但必须记住,较大的数据库将增加运行时间,而较小的数据库如果缺乏相关的蛋白质序列,它们的灵敏度可能会受到影响。对于那些没有针对其特定微生物量身定制的手动策划数据库的人,我们建议使用NCBI-NR(非冗余)蛋白质数据库(或类似数据库,如SwissProt)。
还需要考虑的是,虽然管道使用vanilla BlastP / BlastX运行,但我们已经集成了Diamond,可以调用它,并且会显着减少运行时。--diamond
是Pseudofinder的核心命令。调用此命令将识别输入基因组注释中的假基因候选者,并将生成各种输出文件,下面将详细解释。
打印“annotate”模块的帮助菜单
(Pseudofinder) [user@server ~]# pseudofinder.py annotate -h
运行"annotate"模块
(Pseudofinder) [user@server ~]# pseudofinder.py annotate -g GENOME.gbf -op PREFIX -db /PATH/TO/NR/nr -t 16
"annotate"的输出
| File | Description |
|---|---|
| [prefix]_interactive.html | Interactive plots which summarize the genome-wide analysis. |
| [prefix]_intact.gff | Intact genes in GFF3 format. |
| [prefix]_intact.faa | Intact genes in fasta format. |
| [prefix]_intergenic.fasta | Intergenic regions in fasta format. |
| [prefix]_blastX_output.tsv | Tab-delimited output of BLASTX run on intergenic regions. |
| [prefix]_log.txt | Summary of all inputs, outputs, parameters and results. |
| [prefix]_map.pdf | Concatenated chromosome map. Input genes appear on the inner track in blue, and candidate pseudogenes are shown in red on the outer track. |
| [prefix]_proteome.faa | All protein sequences in fasta format. |
| [prefix]_blastP_output.tsv | Tab-delimited output of BLASTP run on proteome. |
| [prefix]_pseudos.gff | Candidate pseudogenes in GFF3 format. |
| [prefix]_pseudos.fasta | Candidate pseudogenes in fasta format. |
如果包括参考基因组,则运行还将产生:
| File | Description |
|---|---|
| [prefix]_interactive_dnds.html | Interactive genome-wide dN/dS plot. |
| [prefix]_dnds | Directory containing output from the dnds module: BLAST results, dN/dS summary file, and a folder containing the nucleotide, amino acids, and codon alignments that were used to calculate dN and dS values. |
sleuth 命令将一个基因组与另一个密切相关的基因组进行比较。 识别出同源基因后,该模块在比对基因上运行 PAML 以生成密码子比对并计算每个基因的 dN/dS 值。 这些 dN/dS 值可用于推断中性选择和潜在的隐秘假基因。 通过使用 -ref 参数提供密切相关的参考基因组,可以在 Annotate 命令中调用此模块。
用法
# Call within annotate
(Pseudofinder) [user@server ~]# pseudofinder.py annotate -g GENOME.gbf -ref REFERENCE.gbf -op PREFIX -db /PATH/TO/NR/nr -t 16
# Stand alone dN/dS calcuation
(Pseudofinder) [user@server ~]# pseudofinder.py sleuth -a GENOME_PROTS -n GENOME_GENES -ra REFERENCE-PROTS -rn REFERENCE_GENES
Reannotate 将运行注释工作流程,在计算密集型 BLAST 和密码子对齐步骤之后开始。 如果您想更改任何下游参数,此命令可以非常快速地重新注释假基因。 上次运行的日志文件将被解析为以前的参数和文件,因此请将文件保存在日志文件中描述的位置。
用法
(Pseudofinder) [user@server ~]# pseudofinder.py reannotate -g GENOME -log LOGFILE -op OUTPREFIX
Pseudofinder 的优势之一是它能根据用户的需求进行微调。为了可视化更改此程序参数的效果,我们提供了可视化命令。 此命令将显示基于任意组合参数检测到假基因的数量。 与reannotate模块类似,会解析日志文件获取相关文件和参数的信息。
用法
(Pseudofinder) [user@server ~]# pseudofinder.py visualize -g GENOME -log LOGFILE -op OUTPREFIX
只需一个命令,整个 Pseudofinder 工作流程就可以在 Candidatus Tremblaya Princeps strain PCIT 的 139 kbp 基因组上运行(或者,您可以选择提供自己的基因组)。
用法
(Pseudofinder) [user@server ~]# python3 pseudofinder.py test -db /PATH/TO/NR/nr
工作流程将立即开始并将结果写入在./pseudo-finder/test/
- Syberg-Olsen, M. J., Garber, A. I., Keeling, P. J., McCutcheon, J. P., & Husnik, F. (2021). Pseudofinder: detection of pseudogenes in prokaryotic genomes. doi:10.1101/2021.10.07.463580
